单细胞ATAC-seq——肿瘤表观遗传学研究的利器
| 导读 | 尽管目前科学家们的大部分研究是在群体细胞中探寻其相关的表观遗传学平均特征,但是极其复杂和富有异质性的组织更加吸引科学家想“高分辨率”地一探究竟,因此单细胞表观遗传学研究得以受到关注和发展。 |
人类细胞的DNA约有6英尺长,如何将这些DNA装入直径仅为5微米大小的细胞核中是细胞面临的一个巨大的拓扑学挑战。细胞的解决方案是将DNA进行不同层次的折叠形成可以调控的结构,这其中最重要和最基本的结构就是核小体,进一步再由核小体包装形成染色质[1]。
在每个细胞中组成基因组的60亿个碱基中只有小部分具有生物学活性,能与转录元件结合开始合成RNA,而剩下不活跃的DNA则被包装成高度凝聚的染色质而隐藏起来(图1)。因此,染色质像一个“守门员”控制着转录因子、增强子等元件的结合和活性,它不是静态的,而在多个水平上动态变化着[2]。
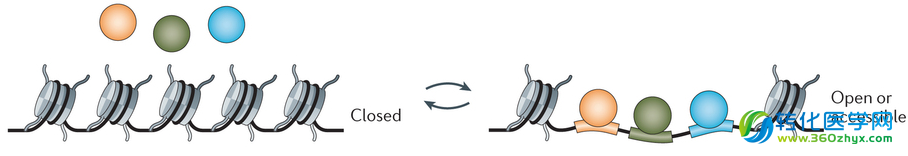
染色质结构的变化需要一系列特殊复合物的协同作用。基因组上这些“开放”或者“可接近性”(accessible) 的区域是转录因子(transcription factors , TFs)和转录元件重要的结合位点,在历史上,一般用它对核酸酶的高敏感性来界定[4]。特别引人注意的是,全基因组范围染色质可接近性分析的方法已经在多种细胞类型中被证明其在调控元件的鉴定过程和度量基因表达的激活及抑制方面极其有效。Boyle和Bernstein等通过绘制群体细胞全基因组范围“开放染色质”的图谱[5]和调控元件的图谱揭示出不同类型细胞的染色质结构的多样性,尤其是在位于远端的调控区域[6]。Thurman等通过对125个不同的细胞和组织进行DHSs(DNase I hypersensitive sites)分析和调控元件注释发现染色质可接近性、转录、DNA甲基化和转录因子对结合位点的占用模式(regulatory factor occupancy patterns)具有不可思议的相关性[7]。
而Buck等通过对比透明细胞性肾细胞癌(clear cell renal cell carcinoma, ccRCC)样本中染色质可接近性分析的数据和DNA-甲基化分析的数据发现有很多位点染色质可接近性下降而DNA-甲基化水平并未发生变化,并且鉴定出一些染色质可接近性下降的区域位于ccRCC相关的基因上,包括PBRM1, SETD2 and MLL2[11],因此,染色质可接近性研究为肿瘤表观遗传学研究中开启了另一扇窗。
目前在基因组范围内染色质可接近性的研究方法很多,例如MNase-seq, FAIRE-seq和DNase-seq[4],这些方法可以帮助我们在大量的细胞系和组织样本中鉴定出转录因子的结合位点、转录活性开始的位置、核小体和核小体修饰、增强子和绝缘子。尽管如此,目前研究染色质可接近性、核小体定位、转录因子占位或者染色质生物学“状态”高级的注释方法一般都涉及多个试验流程,操作复杂,并且需要数万个细胞作为研究部样本,样本准备过程非常繁琐,并且最终得到细胞群体的平均结果。样本量需求如此之大,使得我们没办法用这些方法研究罕见的细胞亚群。
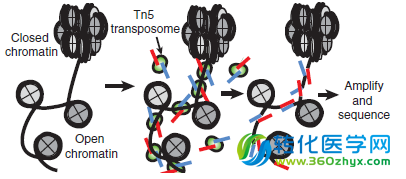
为了解决这些问题,Greenleaf实验室的Buenrostro等发布了一项整合的、多维的遗传学分析方法,这种方法通过Tn5转座酶将测序的adapters插入到基因组上的“可接近”区域来标记调控的区域[9]。这种转座酶可接近性染色质的研究方法(assay of transposase accessible chromatin, ATAC-seq)可以最少只利用大约500个细胞就可以快速的得到调控的多维信息,比其他方法要节省大约3到5个数量级的细胞。并且由于ATAC-seq的实验流程中没有片段选择的步骤,所以这种方法可以同时获得“开放”染色质的位置、转录因子的结合位点、核小体的调控区域和染色质状态等信息[4]。
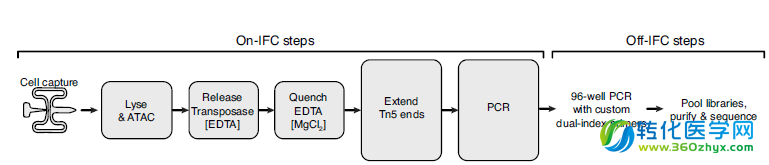
Buenrostro等将ATAC-seq的方法与Fluidigm的单细胞平台C1整合,通过简单的程序利用微流控芯片完成捕获、裂解、转座、PCR等实验过程,建立了自动化的单细胞染色质可接近性图谱研究方法scATAC-seq(Single-cell ATAC-seq)(图4)[10]。

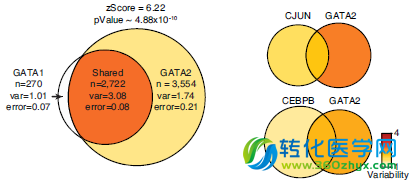
通过对ENCODE中所有可利用的ChIP-seq数据、转录因子结构域和复制期发生变化的染色质区域计算分析,Buenrostro等发现处于不同复制期的结构域染色质可接近性的变异性增加,这与我们对增殖细胞的预期是相同的。同时,Buenrostro等还发现与不同的转录因子可以通过协同或者竞争性结合的作用促进或者抑制染色质可接近性中位点与位点的可变性。例如,GATA1和GATA2这对在K562中异常活跃的转录因子,具有不同的表达水平且结合在相同的序列GATA上,但是同时具有GATA1和GATA2 ChIP-seq信号的调控元件在染色质可接近性的可变性增加,而只结合GATA1或者GATA2的位点则显示较低的可变性(图5A)。而相反的,GATA转录因子在与JUN和CEBPB的协同作用时可变性就不会发生变化。此外,他们的研究还发现相比GATA2,GATA1的结合促使染色质的可接近性增加,是可接近性的催化剂,促使单细胞的染色质的可接近性增加。通过此方法对作者对大量转录因子的ChIP-seq数据研究绘制出了转录因子协同作用改变染色质可接近性的图谱(图5B)。例如,GATA2如果与GATA1、TAL1或者 P300结合在相同的区域将会是染色质可接近性的可变性增加,而CTCF, SUZ12或者 ZNF143一般情况下则对染色质的可接近性变化起到抑制作用。因此,单细胞的可接近性图谱分析可以帮助我们明确转录因子协同作用最终是促进还是抑制了细胞与细胞之间的调控变化。同样的,通过这种方法,Buenrostro等还发现与高可变性相关的转录因子的是细胞类型特异的,在单细胞中染色质状态与组蛋白修饰也与染色质可接近性变化相关[10]。

参考文献
1. ATAC-seq: A Method for Assaying Chromatin Accessibility Genome-Wide
2. Determinants and dynamics of genome accessibility
3 Chromatin organization is a major influence on regional mutation rates in human cancer cells
4. Chromatin accessibility: a window into the genome
5. High-Resolution Mapping and Characterization of Open Chromatin across the Genome
6. ENCODE Project Consortium. An integrated encyclopedia of DNA elements in the human genome.
7. The accessible chromatin landscape of the human genome
8. Transcriptional enhancers: from properties to genome-wide predictions
9. Transposition of native chromatin for fast and sensitive epigenomic profiling of open chromatin, DNA-binding proteins and nucleosome position
10. Single-cell chromatin accessibility reveals principles of regulatory variation
在每个细胞中组成基因组的60亿个碱基中只有小部分具有生物学活性,能与转录元件结合开始合成RNA,而剩下不活跃的DNA则被包装成高度凝聚的染色质而隐藏起来(图1)。因此,染色质像一个“守门员”控制着转录因子、增强子等元件的结合和活性,它不是静态的,而在多个水平上动态变化着[2]。

图 1 染色质的可接近性状态转换[8]
从1879年,W. Flemming提出了染色质(chromatin)这一术语到现今,经过这一个多世纪的研究,人们认识到染色质结构与细胞的生物学调控紧密相连,将影响细胞的转录、复制和修复,与肿瘤产生、发展息息相关。例如,Benjamin Schuster等人通过对多种遗传和表观遗传特征分析发现癌症细胞基因组的基因突变率与染色质组织方式存在显著的相关性。他们的研究发现,在百万碱基数量级上,异染色质相关组蛋白修饰H3K9me3导致40%以上的突变率变化,而多种染色质特性组合将导致55%以上的突变率变化。这种突变率与染色质组织方式强烈的相关性在不同的组织样本和不同类型的突变中得到了印证,暗示着染色质的结构形式很可能是人类体细胞突变率的主要影响因素[3]。因此表观遗传学在短短数年已成为生命科学界最热门领域之一。染色质结构的变化需要一系列特殊复合物的协同作用。基因组上这些“开放”或者“可接近性”(accessible) 的区域是转录因子(transcription factors , TFs)和转录元件重要的结合位点,在历史上,一般用它对核酸酶的高敏感性来界定[4]。特别引人注意的是,全基因组范围染色质可接近性分析的方法已经在多种细胞类型中被证明其在调控元件的鉴定过程和度量基因表达的激活及抑制方面极其有效。Boyle和Bernstein等通过绘制群体细胞全基因组范围“开放染色质”的图谱[5]和调控元件的图谱揭示出不同类型细胞的染色质结构的多样性,尤其是在位于远端的调控区域[6]。Thurman等通过对125个不同的细胞和组织进行DHSs(DNase I hypersensitive sites)分析和调控元件注释发现染色质可接近性、转录、DNA甲基化和转录因子对结合位点的占用模式(regulatory factor occupancy patterns)具有不可思议的相关性[7]。
而Buck等通过对比透明细胞性肾细胞癌(clear cell renal cell carcinoma, ccRCC)样本中染色质可接近性分析的数据和DNA-甲基化分析的数据发现有很多位点染色质可接近性下降而DNA-甲基化水平并未发生变化,并且鉴定出一些染色质可接近性下降的区域位于ccRCC相关的基因上,包括PBRM1, SETD2 and MLL2[11],因此,染色质可接近性研究为肿瘤表观遗传学研究中开启了另一扇窗。
目前在基因组范围内染色质可接近性的研究方法很多,例如MNase-seq, FAIRE-seq和DNase-seq[4],这些方法可以帮助我们在大量的细胞系和组织样本中鉴定出转录因子的结合位点、转录活性开始的位置、核小体和核小体修饰、增强子和绝缘子。尽管如此,目前研究染色质可接近性、核小体定位、转录因子占位或者染色质生物学“状态”高级的注释方法一般都涉及多个试验流程,操作复杂,并且需要数万个细胞作为研究部样本,样本准备过程非常繁琐,并且最终得到细胞群体的平均结果。样本量需求如此之大,使得我们没办法用这些方法研究罕见的细胞亚群。
为了解决这些问题,Greenleaf实验室的Buenrostro等发布了一项整合的、多维的遗传学分析方法,这种方法通过Tn5转座酶将测序的adapters插入到基因组上的“可接近”区域来标记调控的区域[9]。这种转座酶可接近性染色质的研究方法(assay of transposase accessible chromatin, ATAC-seq)可以最少只利用大约500个细胞就可以快速的得到调控的多维信息,比其他方法要节省大约3到5个数量级的细胞。并且由于ATAC-seq的实验流程中没有片段选择的步骤,所以这种方法可以同时获得“开放”染色质的位置、转录因子的结合位点、核小体的调控区域和染色质状态等信息[4]。

图2 ATAC-seq原理图[9]
尽管目前科学家们的大部分研究是在群体细胞中探寻其相关的表观遗传学平均特征,但是极其复杂和富有异质性的组织更加吸引科学家想“高分辨率”地一探究竟,因此单细胞表观遗传学研究得以受到关注和发展。Buenrostro等将ATAC-seq的方法与Fluidigm的单细胞平台C1整合,通过简单的程序利用微流控芯片完成捕获、裂解、转座、PCR等实验过程,建立了自动化的单细胞染色质可接近性图谱研究方法scATAC-seq(Single-cell ATAC-seq)(图4)[10]。

图3 在C1上进行scATAC-seq进行表观遗传研究的试验流程[10]
在实验初始阶段,Buenrostro等用scATAC-seq方法对254个类淋巴母细胞进行了染色质可接近性图谱的分析。他们发现将这些单细胞数据合并分析后得到的结果与用DNase-seq或者ATAC-seq从大约107个细胞中或者104个细胞中得到的可接近性图谱具有很高的相关性(图4)[10],单细胞的数据再现了一些群体细胞ATAC-seq数据反映出的染色质特征。
图4 GM12878 单细胞ATAC-seq数据合并分析后的结果与群体细胞DNase-seq和 ATACseq得到的数据有很高的相关性 [10]
然后他们用scATAC-seq的方法对H1人类胚胎干细胞、K562慢性粒细胞性白血病细胞、GM12878类淋巴母细胞、V6.5小鼠干细胞、EML1细胞(小鼠造血祖细胞)、TF-1细胞(人类成红细胞)、HL-60 cells (人类 promyeloblasts)和BJ成纤维细胞HL-60 细胞这八种类型的细胞进行了染色质可接近性图谱研究。由于单个细胞数据呈现形式的特殊性,Buenrostro等开发了一套专门针对此类数据进行调控变化分析的工具。通过对ENCODE中所有可利用的ChIP-seq数据、转录因子结构域和复制期发生变化的染色质区域计算分析,Buenrostro等发现处于不同复制期的结构域染色质可接近性的变异性增加,这与我们对增殖细胞的预期是相同的。同时,Buenrostro等还发现与不同的转录因子可以通过协同或者竞争性结合的作用促进或者抑制染色质可接近性中位点与位点的可变性。例如,GATA1和GATA2这对在K562中异常活跃的转录因子,具有不同的表达水平且结合在相同的序列GATA上,但是同时具有GATA1和GATA2 ChIP-seq信号的调控元件在染色质可接近性的可变性增加,而只结合GATA1或者GATA2的位点则显示较低的可变性(图5A)。而相反的,GATA转录因子在与JUN和CEBPB的协同作用时可变性就不会发生变化。此外,他们的研究还发现相比GATA2,GATA1的结合促使染色质的可接近性增加,是可接近性的催化剂,促使单细胞的染色质的可接近性增加。通过此方法对作者对大量转录因子的ChIP-seq数据研究绘制出了转录因子协同作用改变染色质可接近性的图谱(图5B)。例如,GATA2如果与GATA1、TAL1或者 P300结合在相同的区域将会是染色质可接近性的可变性增加,而CTCF, SUZ12或者 ZNF143一般情况下则对染色质的可接近性变化起到抑制作用。因此,单细胞的可接近性图谱分析可以帮助我们明确转录因子协同作用最终是促进还是抑制了细胞与细胞之间的调控变化。同样的,通过这种方法,Buenrostro等还发现与高可变性相关的转录因子的是细胞类型特异的,在单细胞中染色质状态与组蛋白修饰也与染色质可接近性变化相关[10]。

图5 (A) GATA1,2相互做用、与JUN、CEBPB协同作用后染色质可接近性变化文氏图

图5(B)转录因子协同作用与染色质可接近性改变图谱
由于真核基因组被精确组装进染色质,所以这些“组装”相关的信息在DNA相关的如转录、DNA修复、复制等生物学过程中占据核心的作用[9],可接近性的差异与特异的反式因子以及顺式作用元件系统性相关。异常的基因表达或者染色质调控因子的改变都将产生深远影响,进而导致各式癌症的产生、发展和转移,而scATAC-seq数据能够帮我们在全基因组范围从多个维度同时揭示关于有关染色质“组装”的重要信息,带我们从单细胞“调控组”的角度探索细胞可塑性和异质性的机制。参考文献
1. ATAC-seq: A Method for Assaying Chromatin Accessibility Genome-Wide
2. Determinants and dynamics of genome accessibility
3 Chromatin organization is a major influence on regional mutation rates in human cancer cells
4. Chromatin accessibility: a window into the genome
5. High-Resolution Mapping and Characterization of Open Chromatin across the Genome
6. ENCODE Project Consortium. An integrated encyclopedia of DNA elements in the human genome.
7. The accessible chromatin landscape of the human genome
8. Transcriptional enhancers: from properties to genome-wide predictions
9. Transposition of native chromatin for fast and sensitive epigenomic profiling of open chromatin, DNA-binding proteins and nucleosome position
10. Single-cell chromatin accessibility reveals principles of regulatory variation
11. Alterations in chromatin accessibility and DNA methylation in clear cell renal cell carcinoma
(转化医学网360zhyx.com)

评论:
共有 1 条评论
-
 游客2017-11-05 16:04:39你这里的插图又不是你们网站画的,为什么要加上自己的水印???!!!
游客2017-11-05 16:04:39你这里的插图又不是你们网站画的,为什么要加上自己的水印???!!!
加载更多
 正在加载
正在加载没有更早的了
 腾讯登录
腾讯登录