如何更好的开展CRISPR研究实现脱靶效应最小化?这里有一份秘籍
| 导读 | 尽管源自化脓链球菌(SpCas9)的第一个且最常用的CRISPR/Cas9系统具有显著的特异性,但除了理想的基因剪切和复制之外,已观察到不同程度的脱靶效应(OTE)。这未免让研究者感到无所适从,由此,如何避免脱靶效应成为了一个重要议题。 |
距离首次作为基因组编辑工具仅仅5年,CRISPR几乎就成为了家喻户晓的技术。从一开始作为细菌切除入侵病毒DNA的方式,到现在,CRISPR已经引发了关于动物模型、高通量筛选、转基因生物和基因治疗,甚至是生物医学伦理学和科学专利法的一系列讨论。
所有的一切都在以令人震撼的速度增长。诚然,CRISPR并非没有缺点,但它的优点太多,涉及到其可编程并适用于无数生物、细胞类型和众多研究。截止2018年,已经发布了超过2500篇CRISPR的论文。可以说CRISPR充满无限的可能,这也许就是它具有让人难以抵抗的魅力所在。
基于此,越来越多的科研人员需要操作CRISPR实验。然而,尽管源自化脓链球菌(SpCas9)的第一个且最常用的CRISPR/Cas9系统具有显著的特异性,但除了理想的基因剪切和复制之外,已观察到不同程度的脱靶效应(OTE)。
这未免让研究者感到无所适从,由此,如何避免脱靶效应成为了一个重要议题,以下是一些CRISPR实验设计的策略,助你实现脱靶效应最小化。
选择合适的靶基因和宿主细胞
在规划基因组编辑实验时,要问的第一个也许是最基本的问题是目的基因是否适合预期的基因编辑操作(即删除、插入或突变)以及特定的细胞环境(细胞类型和细胞系等)。
丨靶基因的选择
首先,确定目标目的基因是否在宿主细胞中表达以及它是否对细胞活力是必需的。虽然已经确定了一组“细胞必需基因”,但有些基因依赖于背景,因此目的基因在特定细胞系中的必需性可能仍需要进一步确定。
其中一个方法是根据siRNA敲低实验的信息,但在某些情况下,细胞可能会在必需蛋白质水平降低的情况下也能存活;此外,小鼠基因敲除(KO)数据的表型读数的也可以检查目的基因的必需性。
然而,通常很难区分生物体发育和细胞存活本身所需基因,一个物种中基因的必需性可能也无法转化为另一个物种的必需性。一些免费的在线工具应运而生,有助于研究者进行相关的分析。
例如,来自华中科技大学、第四军医大学和同济大学等机构的研究人员建立了一个在线分析细胞重要性的OGEE™数据库(http://ogee.medgenius.info./analyze/)。
OGEE™分析界面截图(图片来源:Nucleic Acids Research)
丨细胞的选择
对细胞(原代细胞、hPSC、iPSC或细胞系)信息的了解将有助于确定特定研究的适当实验策略,例如具体操作细胞与研究疾病的生理相关性(组织起源和/或分化状态)。
对于某些多核的细胞类型如骨骼肌细胞和破骨细胞,和携带染色体异常(包括缺失,重复或易位)的许多癌细胞、hPSCs / hESCs /iPSC以及永生化细胞系,使所有等位基因实现有效突变在技术上具有一定的挑战性。
另外,在设计实验方法时,细胞的遗传稳定性是需要考虑的重要因素。由于自发突变和其他基因组影响,特定细胞系的基因组序列不一定与给定基因的公开参考序列相同。因此,应当跨越目的基因区域进行测序以鉴定可能具有干扰gRNA结合或CRISPR结果的序列偏差。
鉴于DNA损伤修复的缺陷是许多形式的人类癌症的基础,源自此类癌症患者的细胞系将积累更多突变并具有更低的基因组稳定性。例如,长期以来报道来源于患有恶性人淋巴细胞个体的细胞系具有比来自正常个体的细胞系更高的突变频率;与来自健康供体的细胞相比,来自患有范可尼贫血和共济失调-毛细血管扩张的患者的B淋巴母细胞样细胞系的突变率分别为30倍和4倍,已知这两种疾病都是由涉及DNA损伤修复的基因突变引起的。
选择合适的宿主细胞系时要考虑的另一个重要方面是它的细胞克隆。虽然许多细胞系最初来源于纯种群(即克隆细胞),但自发突变以及表观遗传变化可能随着培养过程的细胞传代而积累。因此,较低的传代细胞总是优于培养较长时间的细胞。
根据GSK研发人员的经验,阵列比较基因组杂交(aCGH)加上SNP基因组分析是一种简单快捷的确定细胞是否适合用作研究对象的方法。aCGH检测染色体结构和数量的改变,而SNP分析允许检测多倍性、杂合性缺失等。当组合时,这些技术提供关于细胞系同源性和染色体重复/缺失的信息,因此提供关于位于特定区段基因的等位基因拷贝数的信息。此外,还可以对研究中的细胞系进行其他类型的分析,例如STR、FISH和核型分析。
最小化CRISPR脱靶效应的策略
丨适当的gRNA设计
正确选择用于将Cas9核酸酶导向靶基因的指导RNA(gRNA)是在CRISPR基因组编辑中减少OTE的基本步骤之一。
有许多不同的软件工具可用于gRNA的设计,原先大多数程序基于靶DNA选择性对gRNA进行排序以对CRISPR/Cas9的脱靶效应进行预测,但许多较新的程序还包括考虑其他因素的算法,例如目标切割效率、PAM序列等。
目前,可用的gRNA设计工具包括但不限于:
-
DeepCrispr(http://www.deepcrispr.net/)
-
Elevation(https://crispr.ml)
-
Benchling(https://benchling.com/)
-
CRISPRscan(http://www.crisprscan.org/)
-
CHOPCHOP(http://chopchop.cbu.uib.no/)
-
GuideScan(www.guidescan.com)

gRNA设计在线网站
此外,还有支持CRSIPRi/CRISPRa gRNA设计的软件程序,(参考网址:https://portals.broadinstitute.org/gpp/public/analysis-tools/sgrna-design-crisprai
;http://crispr-era.stanford.edu/)。
对于研究者,这么多工具可供选择的同时,可能也会造成选择困难。但通过2种或3种不同的工具,然后选择在每种工具中得分始终较高的gRNA可能是有益的。还有一种选择是,Addgene公司开发的CRISPR Software Matchmaker,它可帮助科学家选择最适合其项目需求的软件(https://blog.addgene.org/the-crispr-software-matchmaker-a-new-tool-for-choosing-the-best-crispr-software-for-your-needs)。

(图片来源:Addgene)
丨核酸酶的选择
核酸酶Cas9能影响切割靶基因的效率和精确度,是限制CRISPR发挥巨大潜力的最主要问题之一,为此科学家设计出了SpCas9的一系列变体。
推荐阅读:科学家找到比Cas9更有效、更安全的酶,能使CRISPR基因编辑工具更好地工作丨医麦猛爆料
最值得注意是SpCas9-HF1、eSpCas9(1.1)和随后设计的超精确变体HypaCas9。这些高保真的SpCas9核酸酶变体,尤其是HypaCas9,在研究中表现出来显著减少的OTE,同时保持与原始版本SpCas9相当的编辑效率。类似地,另一种SpCas9变体“Alt-R S.p. HiFi Cas9”,源自易错PCR诱变的功能筛选,据称具有显著降低的OTE,同时保持了目标效率。
此外,科学家还通过其它策略实现了OTE的降低,包括使用配对的gRNA将切口酶Cas9或dCas9-FokI融合蛋白引导至靶向位点。每种方法都依赖于通过一对gRNA识别两个位置紧密的序列以切割两条DNA链,因此在非靶位点编辑的可能性要低得多。
为了突破PAM序列的限制,其它研究人员使用分子进化设计了一系列的SpCas9的工程化变体,包括xCas9、SaCas9、KKH saCas9等。据报道,这些工程化变体除了具有增加的靶点选择范围外,还保持高靶向效率和特异性。
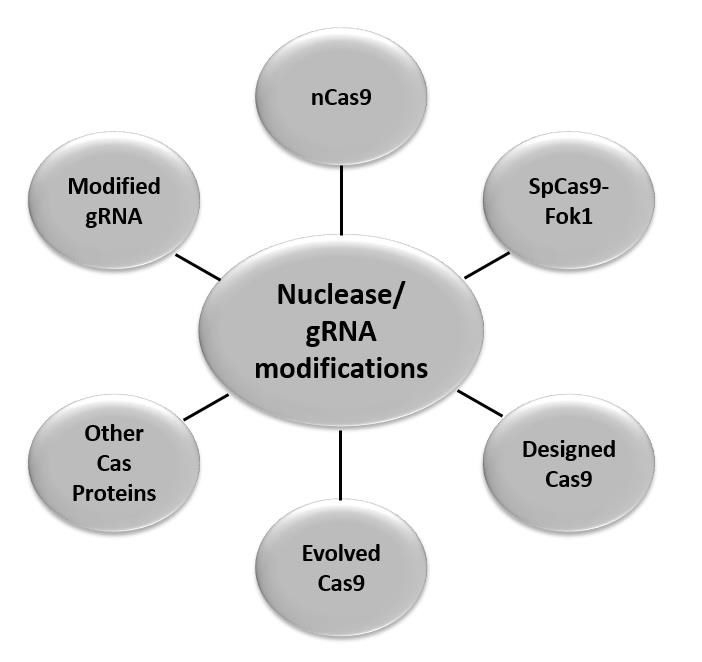
核酸酶的选择(图片来源:Journal of Biotechnology)
丨限制核酸酶的暴露时间
CRISPR核酸酶在宿主细胞内保持活性的时间长短是导致OTE的另一个重要因素。Cas9和gRNA的不同表达水平和持续时间具体取决于所用的递送方法。
在许多情况下,稳定且组成型表达Cas9的细胞系已用于CRISPR实验,虽然Cas9的这种强烈和持续的表达提高了靶基因编辑的效率,但是可能同时增加了OTE,因为Cas9核酸酶的持续和高水平表达促进了gRNA和靶标错配的耐受性。
当慢病毒(LV)用于产生表达Cas9的稳定细胞系时,使用较高感染复数(MOI)将导致更高水平的Cas9表达,但同时也导致更高水平的不期望的病毒整合事件。为了减轻这种影响,Ortinski及其同事设计了一种用于递送Cas9和gRNA基因的整合酶缺陷型慢病毒载体,这促使OTE水平显著降低,同时保持了相当的递送和靶向编辑效率。
除此之外,许多其他方法也可以减少Cas9暴露的持续时间或水平。例如:使用细胞渗透性化合物激活的Cas9融合体,从而通过添加细胞渗透性化合物来稳定CRISPR基因组编辑并且减少OTE;Cas9活性也可以通过使用其天然蛋白质抑制剂来调节,如衍生自单核细胞增生李斯特菌前体的AcrIIA4。
在质粒或病毒表达构建体中,通过Cas9/gRNA核糖核蛋白(RNP)复合物将CRISPR核酸酶递送到宿主细胞中具有显著的优势。据报道,除了与转染DNA或病毒构建体相关的细胞毒性降低,易于调节核酸酶剂量以及节省载体生产时间外,使用RNP能减少OTE,因为其具有快速转换率。而当使用Cas9 mRNA时,也报道了降低的OTE。
然而,使用RNP也可以触发某些细胞中的先天免疫反应,导致CRISPR实验中细胞毒性增加,使用化学修饰或磷酸酶处理的gRNA可以帮助减轻这些反应并增加细胞活力。
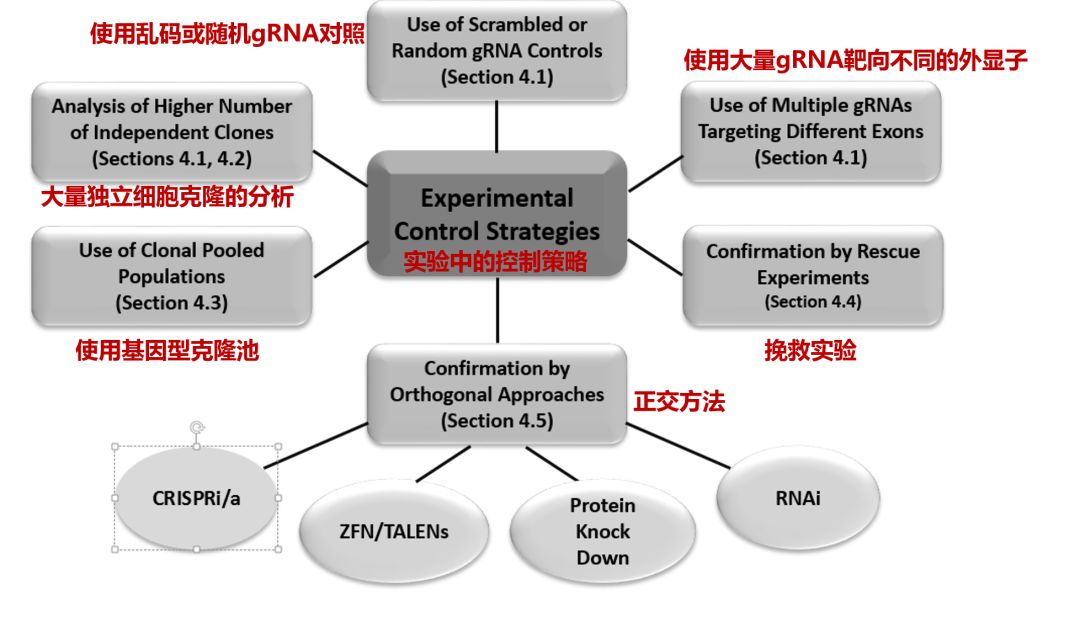
实验设计中控制OTE的策略(图片来源:Journal of Biotechnology)
尽管可以采取措施减少OTE,但现有技术可能无法避免。出于这个原因,研究者不妨在实验设计中采取措施来控制这些效应,以增加对实验结果的信心。
结语
目前,CRISPR基因组编辑技术现在已经超越了实验室,并且正在被开发为有望应用于多种适应症的治疗手段。OTE仍然是牵动CRISPR基因组编辑的临床应用的主要问题。全基因组测序结合传统的安全性评估可能是评估每种治疗剂安全性的金标准。尽管如此,预先确定如何避免OTE的策略将有助于降低开发成本以及提高基于CRISPR的疗法开发的成功率。
上述的体外最小化OTE的策略也可以应用于体内CRISPR基因组编辑。据报道,CRISPR/Cas9基因组编辑后的OTE水平远低于在细胞系中观察到的水平,并且在大多数情况下,当使用当前优化的检测技术时,在测试的动物中没有检测到OTE的证据。
但总是存在引入OTE的机会,并且随着CRISPR/Cas9在治疗用途中的应用,任何OTE都可能对患者产生有害影响。随着该技术更接近于临床环境,我们需要有更稳健、灵敏和有效的方法来确认OTE是否存在,以进一步增强其应用的可信度。
参考出处:
doi: 10.1016/j.jbiotec.2018.08.007
doi: 10.1093/nar/gkw1013
(转化医学网360zhyx.com)
 腾讯登录
腾讯登录
还没有人评论,赶快抢个沙发