【CLIN CHEM】复旦肿瘤医院章真团队:cfDNA片段组学精准预测局晚期直肠癌新辅助放化疗预后
| 导读 | “等待和观察(W&W)”的方法已被用于局部进展期直肠癌(LARC)预后判断,成为了新辅助放化疗(nCRT)后全直肠系膜切除(TME)的替代方案。目前,在判断哪些病人适合使用W&W方法时,主要的判断标准为临床缓完全解(cCR),而非病理完全缓解(pCR)。对于某些患者而言,基于cCR的W&W方式将可能导致比标准根治性手术更差的长期预后。近日,来自复旦大学附属肿瘤医院的研究者突破了这一难题。
|
局部进展期直肠癌(LARC)的标准治疗方法是新辅助放化疗(nCRT)加全直肠系膜切除(TME),并在手术后接受辅助性化疗。然而,此标准疗法虽然可以降低局部进展期直肠癌(LARC)的局部复发率,但也可能增加并发症风险,并对肠道、泌尿和性功能产生长期负面影响。据观察,约有10%-35%接受了nCRT的患者获得了病理完全缓解(pCR);而这些获得了pCR的患者通常具有良好的预后。这意味着某些接受nCRT的病人也许可以免于全直肠系膜切除(TME)。
近年来,一种被称作“等待和观察(W&W)”的方法已被用于LARC预后判断,成为了新辅助放化疗(nCRT)后局部晚期直肠癌(LARC)手术的替代方案,以使患者免于手术切除和生活质量的下降。目前,在判断哪些病人适合使用W&W方法时,主要的判断标准为临床缓完全解(cCR):通过内窥镜检查、影像学检查和体格检查,未在该患者体内检测到残留肿瘤组织的迹象。但是,作为病理完全缓解(pCR)的替代物,临床缓完全解(cCR)并不等同于pCR——对于某些患者而言,基于cCR的W&W方式将可能导致比标准根治性手术更差的长期预后。因此,我们急需对病理完全缓解(pCR)进行预测,以便更准确地筛选哪些患者适合在接受新辅助放化疗(nCRT)后进行“等待和观察(W&W)”。
10月29日,复旦大学上海肿瘤中心章真教授团队在Clinical Chemistry期刊(IF: 12.17)发表了一项大型直肠癌MRD前瞻性临床研究(M-DECIPHER-RC-nCRT),为上述问题提供了答案。该研究证实:较单一影像学特征而言,ctDNA突变和mrTRG联合模型能够准确预测局部进展期直肠癌(LARC)患者在接受新辅助放化疗(nCRT)后的病理完全缓解(pCR)程度。区分pCR和Non-pCR的AUC值高达0.96。

https://academic.oup.com/clinchem/advance-article/doi/10.1093/clinchem/hvac173/6779862?rss=1&login=false
研究背景
01
循环肿瘤DNA(ctDNA)和游离DNA(cfDNA)已被用于预测患者预后、对结直肠癌复发进行早期探测。章真教授团队已在先前的研究中发现:与单独使用MRI相比,结合ctDNA和MRI可以显著提高预测性能;此外,ctDNA还可以识别出被MRI错误分类的患者。然而,一些非pCR病例在接受nCRT治疗后肿瘤负担也会显著下降——在这种情况下,ctDNA和MRI可能都不够灵敏,无法捕获这些病例。
最近,cfDNA片段组学(一种表明肿瘤细胞存在的非遗传特征)已被用于各种癌症的早期检测;这些非遗传特征包括cfDNA片段谱、5′末端基序谱(5′-end motif profiles)。受此启发,章真教授团队将cfDNA片段组学的应用扩展到预测pCR/ non-pCR——这种扩展与癌症检测具有共同的基础。
研究方法
02
此研究招募了119名LARC患者(其中103名患者具有完整的样本),并通过pCR状态和病理或MRI肿瘤消退分级(mrTRG)对nCRT治疗反应进行了评估。研究人员对在接受nCRT前、中、后采集的血浆样本进行了深度靶向测序(deep targeted-panel sequencing)。该103名患者以2:1比例被随机分为训练集(n=68)和验证集(n=35)。cfDNA片段和5′末端基序图谱用于构建弹性网络逻辑回归模型(elastic-net logistic regression models)以预测非pCR。通过AUC(Area Under Curve)、灵敏度和特异性来衡量该模型的预测性能。
Workflow of the study:
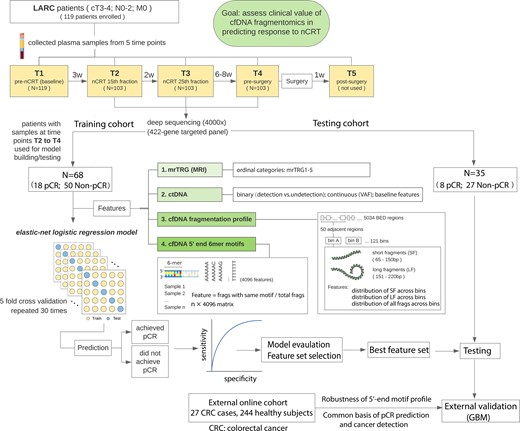
研究结果
03
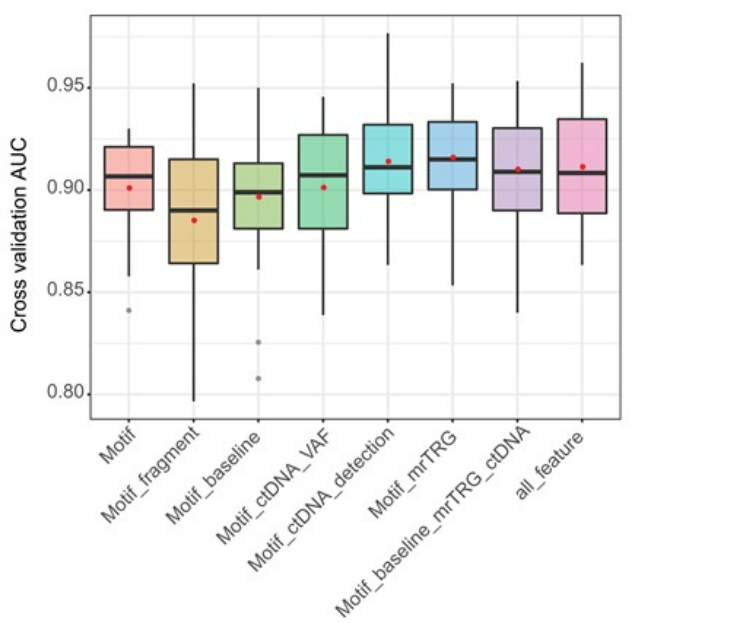
广谱特征用于pCR预测表现出优良性能:不管是Motif还是Fragment,均显示出较好的nCRT疗效预测能力,模型交叉验证AUC分别是0.90(95%CI: 0.89-0.91)和0.86(95%CI: 0.85-0.87)。Motif与Fragment的广谱特征对nCRT疗效预测具有重要作用。
末端基序与mrTRG的联合模型较单一特征显著提高pCR预测性能:此研究联合了cfDNA广谱特征、ctDNA突变特征和影像学特征,进一步探索了pCR预测性能的提升。T234_Motif和mrTRG联合模型区分pCR和Non-pCR,AUC高达0.92(95%CI: 0.91-0.93),显著优于单一特征和其他特征组合。在独立验证集中,该模型的预测性能同样优异,AUC高达0.96(95%CI: 0.90-1.00)。
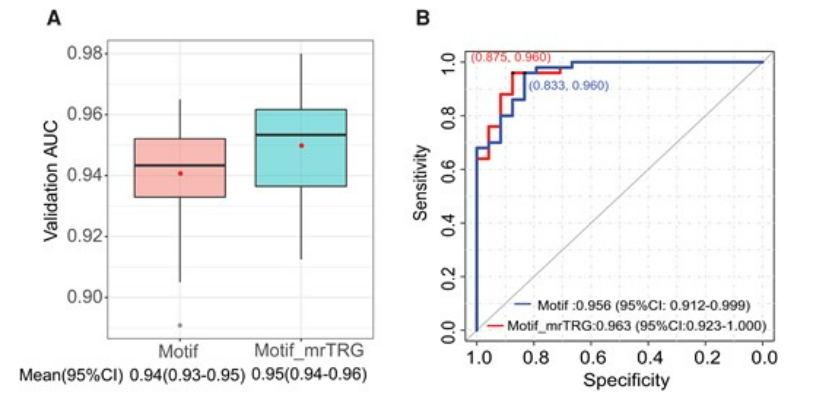
联合模型在ctDNA-MRD阴性患者中仍可展现优异性能:在74例nCRT治疗期间外周血无法检测到ctDNA突变的患者中,Motif特征或Motif与mrTRG的联合模型仍然表现出了良好的pCR预测能力;其AUC分别是0.94(95%CI: 0.93-0.95)和0.95(95%CI: 0.94-0.96)。这些结果表明cfDNA广谱特征及其联合模型可独立于突变特征来对pCR进行预测。
此外,此研究还使用当地队列作为训练队列,其中pCR对应于健康,非pCR对应于癌症。27名CRC患者和244名健康受试者组成了外部验证队列。GBM模型在训练集和外部独立验证集中的AUC分别为0.885(95%CI: 0.804-0.965)和0.878(95%CI: 0.801-0.956),预测性能优异。该结果表明了了基于cfDNA末端基序的广谱模型稳健性。
研究意义
04
在该研究中,对于预测non-pCR ,cfDNA片段谱和5'末端基序谱都显示出较高的样本外验证AUC值。值得一提的是,对于无法检测到ctDNA的患者,5'末端基序谱仍然保持着良好的预测能力 ;支持了5'末端基序谱在pCR/非pCR预测和癌症早期检测中的普遍应用。该研究将多组学技术应用于围术期MRD监测,建立了局部进展期直肠癌新辅助pCR预测模型,进而更为精准地筛选出适合接受“等待和观察(W&W)”的LARC患者,为LARC的新辅助治疗预后判断提供了精准参考。(转化医学网360zhyx.com)
参考资料:
https://academic.oup.com/clinchem/advance-article/doi/10.1093/clinchem/hvac173/6779862?rss=1&login=false
https://www.genomeweb.com/scan/study-incorporates-circulating-tumor-fragments-rectal-cancer-treatment-response-models#.Y2H742lBxdh
注:本文旨在介绍医学研究进展,不能作为治疗方案参考。如需获得健康指导,请至正规医院就诊。
 腾讯登录
腾讯登录

还没有人评论,赶快抢个沙发