lncRNA可在癌症诊断与预后中作为生物标志物
| 导读 | 为了增加对癌症发病机理的研究,癌症和肿瘤基因图谱计划(The Cancer Genome Atlas, TCGA)通过高通量测序方案对遗传学,表观遗传学以及转录变化进行了大规模研究。 |

癌症已经成为21世纪世界性的威胁人类健康与安全的头号杀手。2012年就有超过1200万人被确认为新的癌症患者,超过800万人因癌症而死亡[1]。为了增加对癌症发病机理的研究,癌症和肿瘤基因图谱计划(The Cancer Genome Atlas, TCGA)通过高通量测序方案对遗传学,表观遗传学以及转录变化进行了大规模研究[2,3]。然而,尽管很多证据证明了很多基因组非编码区同样可以产生功能性的RNA,人们对此的关注焦点却还是主要局限于蛋白编码的基因上[4]。lncRNA是一类长度大于200个碱基的具有生物学功能的非编码RNA,与编码基因类似的是,lncRNA可以以染色体重组(Chromatin reprogramming),增强子顺式元件(cis regulation at enhancers)以及转录后调控(Post-trtanscriptional regulation)的方式,广泛的参与生物、发育以及病理过程[5,6]。
高通量测序技术的出现为系统性的寻找转录调控元件起到了革命性的推动作用。不仅如此,高通量测序还在错综复杂的天然基因组环境中鉴定lncRNA同样提供了有效的帮助[4]。RNA-seq中比对到的数据经过拼接已经成功的鉴定到了多个新的与癌症有关的lncRNA[7]。部分lncRNA被发现在很多肿瘤组织的发生、扩展以及扩散中存在反常的高表达。因此,这些lncRNA也被认为和癌变或者肿瘤的抑制存在一定的关联,其表达水平的高低意味着预后的好坏。正因为这些lncRNA在癌组织中的特殊特性,其也被认为是潜在的可以预兆组织癌变的生物标志物。
SChLAP1作为生物标志物的研究
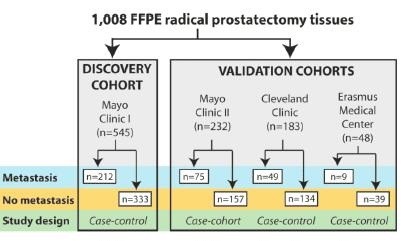
很多疾病的产生都会伴随着细胞或组织中某些生化指标的改变,如分子表达量的变化等。那些可以标记系统、器官、组织、细胞及亚细胞结构或功能的改变或可能发生的改变的生化指标被称为生物标志物。对生物标志物的开发与应用在疾病诊断、判断疾病分期以及新药的研发中起到了重要的作用。2014年12月,国际著名癌症期刊杂志Lancet Oncology(影响因子24.69)发表了一篇名为Nomination and validation of the long noncoding RNA SChLAP1 as a risk factor for metastatic prostate cancer progression: a multi-institutional high-throughput analysis的论文[8]。该论文中,作者分析了1008个前列腺癌(Prostatic cancer)组织样本,确认了其中一个名为SChLAP1的lncRNA(图1)。该lncRNA特异的出现在前列腺癌细胞中,并且由于在扩散的前列腺癌细胞中表达量远高于前期的前列腺癌细胞而受到关注。作者认为该lncRNA在前列腺癌细胞中的特异表达可以作为一个有效的生物标志物应用于对前列腺癌的确认以及术后观察。
HOTAIR同样也是一个在癌症研究中被认为和癌症的发生有密切关系的lncRNA[9]。该lncRNA首先被Gupta等人在扩散的乳腺癌(Breast cancer)细胞中鉴定到[10]。肿瘤中高表达的HOTAIR意味着癌症的晚期以及死亡[10]。在乳腺癌细胞中过表达HOTAIR会改变H3K27的甲基化状态,诱导癌细胞更具有扩散能力。然而,有趣的是,另一个乳腺癌抑制因子BRCA1可以通过竞争性的结合PRC2的EZH2亚基从而抑制HOTAIR对PRC2的依赖性,进而抑制乳腺癌细胞的发展[11]。
HOTAIR作为一个致癌因子,同样在肝癌细胞(Hepatocellular carcinoma)等癌组织中存在[12,13]。HOTAIR在肿瘤细胞中的高表达往往意味着癌症的复发风险增大以及肝脏切除的可能性增加以及癌细胞向淋巴结(Lymph node)转移[13]。因此,可以认为HOTAIR的高表达对于很多癌症治疗而言是一个负面的生物标记物,这些癌症包括了胰腺癌(Pancreatic cancer)[14]、肺癌(Lung cancer)[15,16]、食道癌(Esophageal carcinoma)[17]、宫颈癌(Cervical cancer)[18]以及结肠癌(Colon cancer)[19]。针对大量消化道恶性癌症存活病人的分析发现,HOTAIR是一个在癌症诊断和预后检测中非常可靠的生物标志物[20]。
生物标志物的筛选
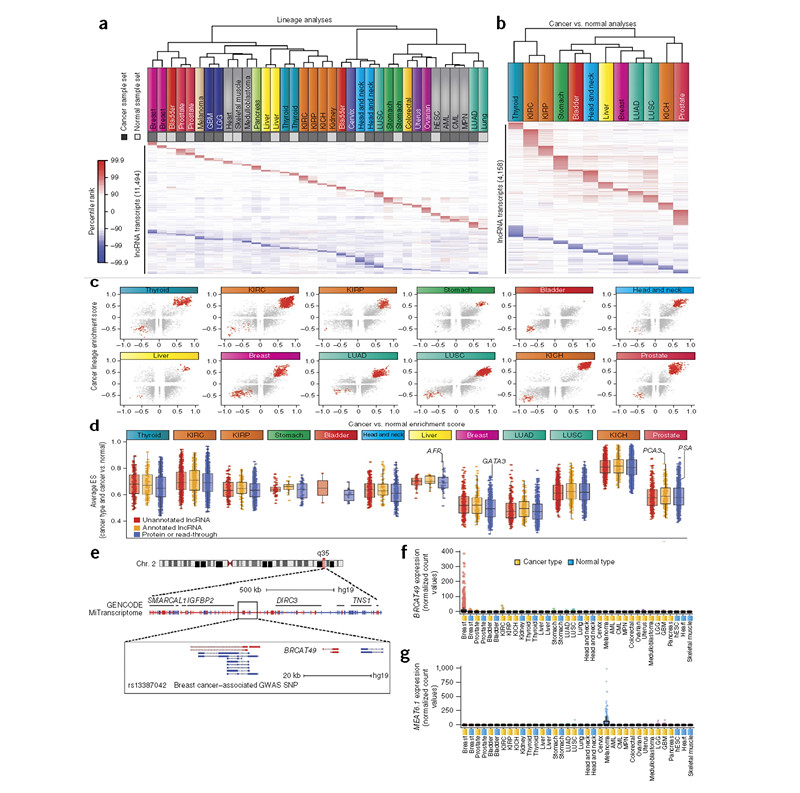
lncRNA作为一类新鉴定到的RNA分子,作为生物标志物的应用远不止于此。目前,已有大量的lncRNA被鉴定为癌症的生物标志物(表一)[21]。Kiyer等更是在对7256个肿瘤样本、正常组织样本以及细胞系总共43Tb数据量的RNA-seq数据进行分析后发现,有7%(大约3900个)的lncRNA与疾病相关的SNPs重叠。通过对全部lncRNA的非参数差异表达分析(non-parametric differential expression testing),作者认定了7942个与癌症有关的lncRNA,并且认为有可能能够成为特定癌组织的生物标志物(图2)。该成果被发表在了2014年6月的Nature Genetics(影响因子29.352)上[22]。
|
lncRNA |
癌症种类 |
致癌性 |
生物标志物含义 |
|
HOTAIR |
乳腺癌 |
致癌 |
癌细胞转移/存活率低 |
|
|
肝癌 |
致癌 |
癌细胞再生/存活率低 |
|
|
结肠/直肠癌 |
致癌 |
癌细胞转移/存活率低 |
|
|
胃癌 |
致癌 |
淋巴结转移 |
|
|
胰腺癌 |
致癌 |
存活率低 |
|
|
肺癌 |
致癌 |
癌细胞转移/存活率低 |
|
|
食道癌 |
致癌 |
癌细胞转移/存活率低 |
|
|
宫颈癌 |
致癌 |
癌细胞转移/存活率低 |
|
MALAT1 |
肺癌 |
致癌 |
癌细胞转移/存活率低 |
|
|
肝癌 |
致癌 |
转移与复发 |
|
|
结肠/直肠癌 |
致癌 |
术后恢复差 |
|
|
宫颈癌 |
致癌 |
|
|
|
膀胱癌 |
致癌 |
癌细胞转移 |
|
|
子宫癌 |
致癌 |
|
|
|
骨肉瘤 |
致癌 |
化疗效果差 |
|
H19 |
食道癌 |
致癌 |
|
|
|
乳腺癌 |
致癌 |
|
|
|
肺癌 |
致癌 |
|
|
|
膀胱癌 |
致癌 |
|
|
|
卵巢癌 |
致癌 |
|
|
|
宫颈癌 |
致癌 |
|
|
|
骨肉瘤 |
致癌 |
|
|
|
颈部鳞癌 |
致癌 |
|
|
|
肝癌 |
致癌 |
|
|
ncRAN |
神经细胞瘤 |
致癌 |
存活率低 |
|
|
膀胱癌 |
致癌 |
|
|
|
结肠/直肠癌 |
肿瘤抑制因子 |
存活率低 |
|
HULC |
肝癌 |
致癌 |
|
|
GAS5 |
乳腺癌 |
肿瘤抑制因子 |
|
|
|
肾癌 |
肿瘤抑制因子 |
|
|
|
胰腺癌 |
肿瘤抑制因子 |
|
|
|
膀胱癌 |
肿瘤抑制因子 |
|
|
|
肺癌 |
肿瘤抑制因子 |
|
|
|
胃癌 |
肿瘤抑制因子 |
预后差 |
|
|
弥漫性恶性间皮瘤 |
肿瘤抑制因子 |
预后差 |
|
|
肝癌 |
肿瘤抑制因子 |
预后差 |
|
PCA3 |
前列腺癌 |
致癌 |
尿液/血液无创诊断 |
|
UCA1 |
乳腺癌 |
致癌 |
尿液无创诊断 |
|
PCGEM1 |
前列腺癌 |
致癌 |
患癌风险大 |
|
PRNCR1 |
前列腺癌 |
致癌 |
患癌风险大 |
|
MEG3 |
白血病 |
肿瘤抑制因子 |
CpG甲基化存活率低 |
|
|
垂体瘤 |
肿瘤抑制因子 |
|
|
|
脑膜瘤 |
肿瘤抑制因子 |
癌细胞扩散能力下降 |
|
|
神经胶质瘤 |
肿瘤抑制因子 |
|
|
NBAT-1 |
神经细胞瘤 |
肿瘤抑制因子 |
癌细胞扩散能力下降 |
|
LincRNA-ROR |
肝癌 |
致癌 |
癌细胞化疗抵抗能力增强 |
|
PCAT1 |
前列腺癌 |
致癌 |
癌细胞扩散 |
尽管中心法则依然是细胞生物学与分子生物学的主流思想,lncRNA作为有生物学功能的非编码RNA已经对中心法则的准确性与全面性产生了重大的影响。其生物学功能在生物学与疾病的研究中也广泛被报道。综合目前已有的报道,lncRNA不同在生物中种类数量巨大,其中部分已经确认与肿瘤发生和癌症形成密切相关。随着人们对lncRNA研究的深入,会有更多的lncRNA被用于生物标志物的检测与开发中,为人类攻破癌症提供有效的帮助。
参考文献
1.Ferlay, J. et al. Cancer incidence and mortality worldwide: Sources, methods and major patterns in GLOBOCAN 2012. International Journal of Cancer 136, E359-E386 (2015).
2.Cyriac, K. et al. Mutational landscape and significance across 12 major cancer types. Nature 502, 333-339 (2013).
3.Ciriello, G. et al. Emerging landscape of oncogenic signatures across human cancers. Nature genetics 45, 1127-1133 (2013).
4.Sarah, D. et al. Landscape of transcription in human cells. Nature 489, : 101-108. (2012).
5.Ulitsky, I. & Bartel, D. P. lincRNAs: Genomics, Evolution, and Mechanisms. Cell 154, 26-46 (2013).
6.Prensner, J. R. & Chinnaiyan, A. M. The emergence of lncRNAs in cancer biology. Cancer Discovery 1, 391-407 (2011).
7.Prensner, J. R. et al. Transcriptome sequencing across a prostate cancer cohort identifies PCAT-1, an unannotated lincRNA implicated in disease progression. Nature Biotechnology 29, 742-749 (2011).
8.Zhao, S. et al. Identification and Validation of the Long Noncoding RNA SChLAP1 as a Prognostic Biomarker in Prostate Cancer. International journal of radiation oncology, biology, physics 90, S2 (2014).
9.Wu, Y. et al. Long noncoding RNA HOTAIR involvement in cancer. Tumour Biology the Journal of the International Society for Oncodevelopmental Biology & Medicine 35, 9531-9538 (2014).
10.Gupta, R. A. et al. Gupta RA, Shah N, Wang KC et al.Long non-coding RNA HOTAIR reprograms chromatin state to promote cancer metastasis. Nature 464:1071-1076. Nature 464, 1071-1076 (2010).
11.Lan, W. et al. BRCA1 is a negative modulator of the PRC2 complex. Embo Journal 32, 1584–1597 (2013).
12.Geng, Y. J., Xie, S. L., Li, Q., Ma, J. & Wang, G. Y. Large intervening non-coding RNA HOTAIR is associated with hepatocellular carcinoma progression. Journal of International Medical Research 39, 2119-2128 (2011).
13.Zhe, Y. et al. Overexpression of Long Non-coding RNA HOTAIR Predicts Tumor Recurrence in Hepatocellular Carcinoma Patients Following Liver Transplantation. Annals of Surgical Oncology 18, 1243-1250 (2011).
14.Kim, K., . et al. HOTAIR is a negative prognostic factor and exhibits pro-oncogenic activity in pancreatic cancer. Oncogene 32, 1616-1625 (2013).
15.Xiang-Hua, L. et al. The long non-coding RNA HOTAIR indicates a poor prognosis and promotes metastasis in non-small cell lung cancer. Bmc Cancer 13, 1-10 (2013).
16.Nakagawa, T. et al. Large noncoding RNA HOTAIR enhances aggressive biological behavior and is associated with short disease-free survival in human non-small cell lung cancer. Biochemical & Biophysical Research Communications 436, 319-324 (2013).
17.Xiao-Bin, L. et al. Long Noncoding RNA HOTAIR Is a Prognostic Marker for Esophageal Squamous Cell Carcinoma Progression and Survival. Plos One 8, e63516 (2013).
18.S, W. et al. Overexpression of long non?coding RNA HOTAIR predicts a poor prognosis in patients with acute myeloid leukemia. Oncology Letters 10 (2015).
19.Ze-Hua, W. et al. Long non-coding RNA HOTAIR is a powerful predictor of metastasis and poor prognosis and is associated with epithelial-mesenchymal transition in colon cancer. Oncology Reports 32, 395-402 (2014).
20.Shenghong, Z. et al. Long noncoding RNA HOTAIR as an independent prognostic marker in cancer: a meta-analysis. Plos One 9, e105538-e105538 (2014).
21.Yarmishyn, A. A. & Kurochkin, I. V. Long noncoding RNAs: a potential novel class of cancer biomarkers. Frontiers in genetics 6, 145.
22.Iyer MK, Niknafs YS, Malik R, Singhal U, Sahu A, et al. (2015) The landscape of long noncoding RNAs in the human transcriptome. Nat Genetics 47: 199-208.
伯豪生物可为您提供lncRNA芯片及测序服务,
 腾讯登录
腾讯登录
还没有人评论,赶快抢个沙发