lncRNA测序在非编码RNA研究中的应用案例
| 导读 | lncRNA测序是通过rRNA去除法从而对含有polyA尾和不含有polyA尾的RNA一同富集建库测序。其优势在于相比于普通的转录组测序而言,lncRNA 测序还可以对那些不含有polyA尾的非编码RNA进行测序,从而鉴定到更多的lncRNA。 |
在过去几年中,高通量转录组测序(High-throughput transcriptome sequencing)最大的发现之一莫过于在哺乳动物基因组中发现了一些功能各不相同的RNA[1]。除去那些由于转录起始位点(Transcriptional start sites)的改变以及终止子(Ternination)和可变剪切(Splicing)的变化之外,另一些新的反向序列(Antisense),基因内(Intronic)以及基因间(Intergenic)的转录本也被陆续发现。其中,超过一半的含有polyA尾的RNA由于不具备编码蛋白的功能,被称为非编码RNA(Non-protein-coding RNAs,ncRNA),这些ncRNA在细胞调控,发育以及疾病的发展过程中起到了重要作用[2]。
在这些ncRNA中,碱基长度大于200bp的又被称为长链非编码RNA(lncRNA)。近些年的研究发现,lncRNA远非曾经想象中的垃圾序列,其在生命体中承担了极其重要的功能,广泛参与了基因沉默[3],X染色体失活[4]以及发育[5-7]。然而,和lncRNA在生命体中广泛的生物学功能相比,人们对它的认识却还是少之又少的。在人们对lncRNA的研究中,无论是新lncRNA的筛选还是lncRNA的功能分析,lncRNA测序都在其中发挥了重要的作用[8-10]。lncRNA测序也称为全转录组测序,与普通的通过polyA富集方法的转录组测序不同的是,lncRNA测序是通过rRNA去除法从而对含有polyA尾和不含有polyA尾的RNA一同富集建库测序。其优势在于相比于普通的转录组测序而言,lncRNA 测序还可以对那些不含有polyA尾的非编码RNA进行测序,从而鉴定到更多的lncRNA。该优势也正使得lncRNA测序更多的在目前的lncRNA研究中被使用。
lncRNA测序筛选新lncRNA
Genome-wide identification of novel long non-coding RNAs in Populus tomentosa tension wood, opposite wood and normal wood xylem by RNA-seq [11].
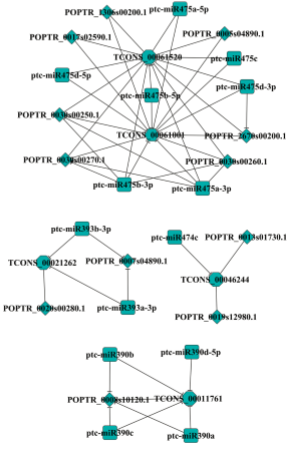
2015年,北京林业大学生物科学与技术学院张德强教授在植物学杂志Planta(影响因子3.263)上发表了一篇毛白杨的lncRNA测序的文章。作者为了了解lncRNA在毛白杨木材形成过程中的作用,使用了上海伯豪的lncRNA测序服务对木质部的NW(Normal wood),TW(Tension wood)以及OW(Opposite wood)组织分别进行了测序,筛选lncRNA。并且作者还对筛选出的lncRNA进行了顺式(-cis)以及反式(-trans)靶标基因的筛选以及GO分类,确认了lncRNA在毛白杨木质化生物学过程中起到了重要作用(图1,2)。
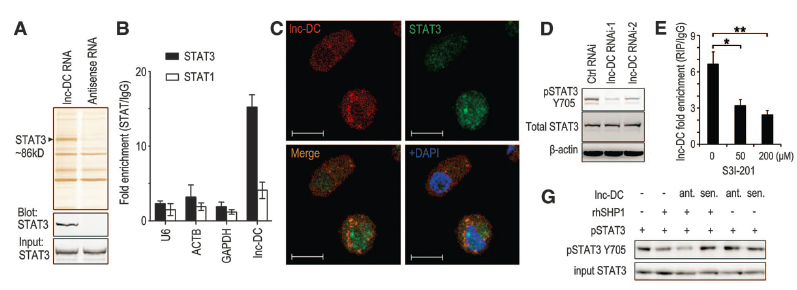
案例一:The STAT3-Binding long noncoding RNA lnc-DC controls human dendritic cell differentiation [12].
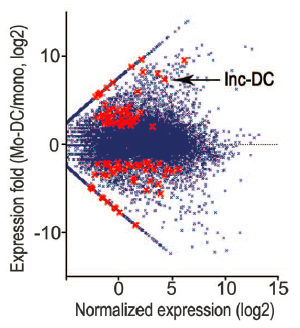
第二军医大学免疫学研究所所长,中国医学科学院院长,兼浙江大学免疫学研究所所长曹雪涛院士在2014年4月18日发表的国际著名期刊Science杂志(影响因子33.611)上发表了一篇介绍lncRNA在人类树突状细胞(Human dendritic cell,hDC)分化上的具体作用与功能。在该文章中,作者鉴定到了一个在人类树突状细胞中高表达的,被命名为lnc-DC的长链非编码RNA。该lncRNA可破坏体外人类外周血单核细胞(Peripheral blood monocytes)以及体内的小鼠骨髓细胞分化为 DCs,并减小了 DCs 刺激T细胞激活的能力。进一步的机制研究发现,lnc-DC发挥其生物学功能是通过激活转录因子STAT3来实现的。lnc-DC直接结合到细胞质中的STAT3上,通过阻止STAT3结合SHP1及被去磷酸化,促进了STAT3 tyrosine-705位点磷酸化(图2)。其中,作者为了确认在DC细胞分化过程中有表达的lncRNA,使用了lncRNA测序对外周血单核细胞进行了lncRNA的高通量测序实验,通过该实验方法,作者成功鉴定到了长链非编码RNA--lnc-DC(图3)。该工作表明,lncRNA可以通过直接与胞质中信号分子的相互作用影响细胞分化,并且调控其转录后修饰。lnc-DC作为一个DC分化过程中特异的调控因子,可以在DC功能紊乱的治疗中具备一定的应用潜力。
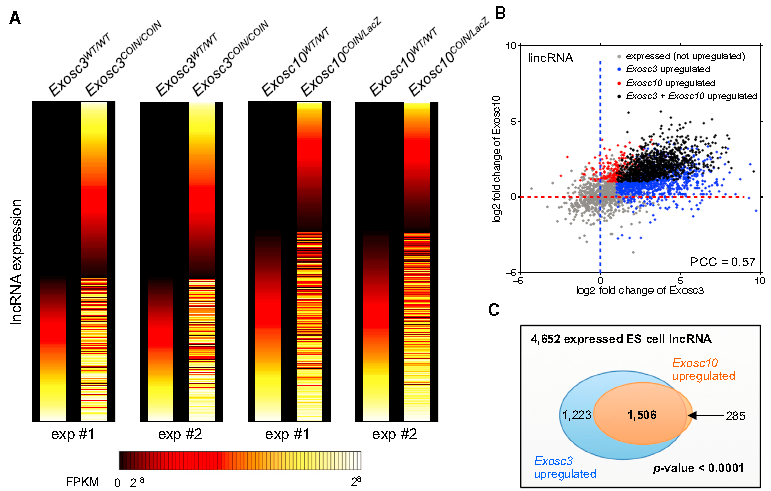
除了上文中通过测序来鉴定感兴趣的lncRNA外,lncRNA测序还可以对某个基因突变后全细胞范围内lncRNA表达产生的变化进行评估。来自于哥伦比亚大学的团队便通过对胚胎干细胞(ESCs)和B细胞的RNA复合体(RNA exosome)中两个重要的突变体Exosc10和Exosc3进行了lncRNA测序(图3)。作者发现,这两个基因敲除的突变体中,大量lncRNA,反向RNA以及增强RNA(eRNA)的表达水平均出现大幅提高。对这些RNA的转录本富集程度的观察发现,这些非编码RNA的稳定性得到了显著增强。lncRNA测序实验确定了Exosc10与Exosc3在RNA复合物介导的lncRNA降解途径中的重要作用。不仅如此,作者还通过对lncRNA测序获得的数据分析后发现,大部分的lncRNA(以Hoxa1为例)同时受到Exosc10与Exosc3这两个亚基的调控。
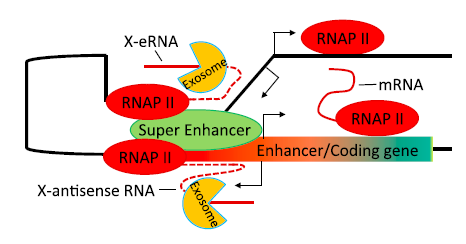
在之后的功能分析中,作者又先后发现了RNA复合物与eRNA的结合,并且通过对eRNA的降解来抑制DNA/RNA复合物的形成或者促进染色体向异染色质的转变。这样的功能可以减少细胞在基因组不稳定时所受到的影响,保护细胞不受损害。
最后,作者还提出了一个lncRNA介导的RNA复合物/超级增强子理论为基因表达调控提供了一个新的模型(图4)。该成果被发表在了2015年3月7日国际著名学术期刊Cell杂志(影响因子32.242)上。
lncRNA测序作为一种比较新颖的高通量测序实验方法,其相对传统转录组测序的优势已经越来越被人们所认可,在基因筛选与功能分析上也已经做出了重要的贡献。随着人们对lncRNA研究的逐步深入,相信lncRNA测序在lncRNA的研究中能够有更多的发现。
参考文献
1 Cesana, M. et al. A Long Noncoding RNA Controls Muscle Differentiation by Functioning as a Competing Endogenous RNA. Cell 147, 358-369 (2011).
2 Sigova, A. A. et al. Divergent transcription of long noncoding RNA/mRNA gene pairs in embryonic stem cells. Proc Natl Acad Sci USA 110, 2876–2881 (2013).
3 Wenqiang, Y. et al. Epigenetic silencing of tumour suppressor gene p15 by its antisense RNA. Nature 451, 202-206 (2008).
4 Zhao, J., Sun, B. K., Erwin, J. A., Song, J. J. & Lee, J. T. Polycomb Proteins Targeted by a Short Repeat RNA to the Mouse X Chromosome. Science 322, 750-756 (2008).
5 Young, T. L., Matsuda, T. & Cepko, C. L. The noncoding RNA taurine upregulated gene 1 is required for differentiation of the murine retina. Current Biology Cb 15, 501-512 (2005).
6 Rinn, J. L., Bondre, C., Gladstone, H. B., Brown, P. O. & Chang, H. Y. Anatomic Demarcation by Positional Variation in Fibroblast Gene Expression Programs. Plos Genetics 2, e119 (2006).
7 Jae Bok, H. & Sibum, S. Vernalization-Mediated Epigenetic Silencing by a Long Intronic Noncoding RNA. Science 331, 76-79 (2011).
8 Liu, J. et al. Genome-wide analysis uncovers regulation of long intergenic noncoding RNAs in Arabidopsis. The Plant cell 24, 4333-4345.
9 Niazi, F. & Valadkhan, S. Computational analysis of functional long noncoding RNAs reveals lack of peptide-coding capacity and parallels with 3' UTRs. Rna 18, 825-843.
10 Klattenhoff, C. A. et al. Braveheart, a long noncoding RNA required for cardiovascular lineage commitment. Cell 152, 570-583.
11 Chen, J., Quan, M. & Zhang, D. Genome-wide identification of novel long non-coding RNAs in Populus tomentosa tension wood, opposite wood and normal wood xylem by RNA-seq. Planta 241, 1-19 (2015).
12 Wang, P. et al. The STAT3-binding long noncoding RNA lnc-DC controls human dendritic cell differentiation. Science 344, 310-313.
13 Pefanis, E. et al. RNA Exosome-Regulated Long Non-Coding RNA Transcription Controls Super-Enhancer Activity. Cell 161, 774-789.
(转化医学网360zhyx.com)
 腾讯登录
腾讯登录
还没有人评论,赶快抢个沙发