一篇 Nature 文吓坏了科研人,难道 CRISPR 脱靶没救了?
| 导读 | 后续调查结果发现,所谓大规模的脱靶现象是作者使用了基因背景不明确的小鼠对照而产生的错误判断,Nature Methods 杂志最终单方面决定,在通讯作者全都不同意的情况下强行撤稿这篇有争议的论文,打破了学术界的惯例。 |
2017 年 5 月发表在 Nature Methods 杂志上,题为「Unexpected mutations after CRISPR-Cas9 editing in vivo」的研究轰动了整个学术圈。该文章声称 CRISPR-Cas9 基因编辑系统会引入大量不可预估的突变到小鼠基因组中,由此引发了关于 CRISPR-Cas9 系统潜在脱靶效应的一系列激烈的讨论。后续调查结果发现,所谓大规模的脱靶现象是作者使用了基因背景不明确的小鼠对照而产生的错误判断,Nature Methods 杂志最终单方面决定,在通讯作者全都不同意的情况下强行撤稿这篇有争议的论文,打破了学术界的惯例[1]。

图 1. 该文章在 2018 年 3 月被正式撤稿[2]
虽然这场让研究人员们「草木皆兵」的风波暂时平息,但也从侧面反映出 CRISPR-Cas9 的主要限制:不仅在其目标序列切割,还对相似序列进行切割。而脱靶切割可能发生在整个基因组中,导致产生潜在的突变及危害。这对 CRISPR 的各种应用,尤其是临床上遗传疾病或癌症治疗提出了尖锐的挑战。本文将重点介绍检测 CRISPR 脱靶效应的方法和应对策略,希望助广大 CRISPR 领域研究者一臂之力。
01
算法预测验证
CRISPR 脱靶效应的关键因素之一是向导 RNA(gRNA),即结合引导 Cas9 核酸酶在基因组上特定位置切割的 RNA 序列。在设计 gRNA 时,我们可通过特定算法预测潜在脱靶位点,尽可能的选择脱靶效应低的 gRNA。在基因组切割之后再进行靶向测序,验证这些位点突变情况,以评估脱靶效应。
现在已有基于各种算法的预测工具,总结详见表 1:
表 1. gRNA 设计和脱靶效应分析工具
(点击查看大图)
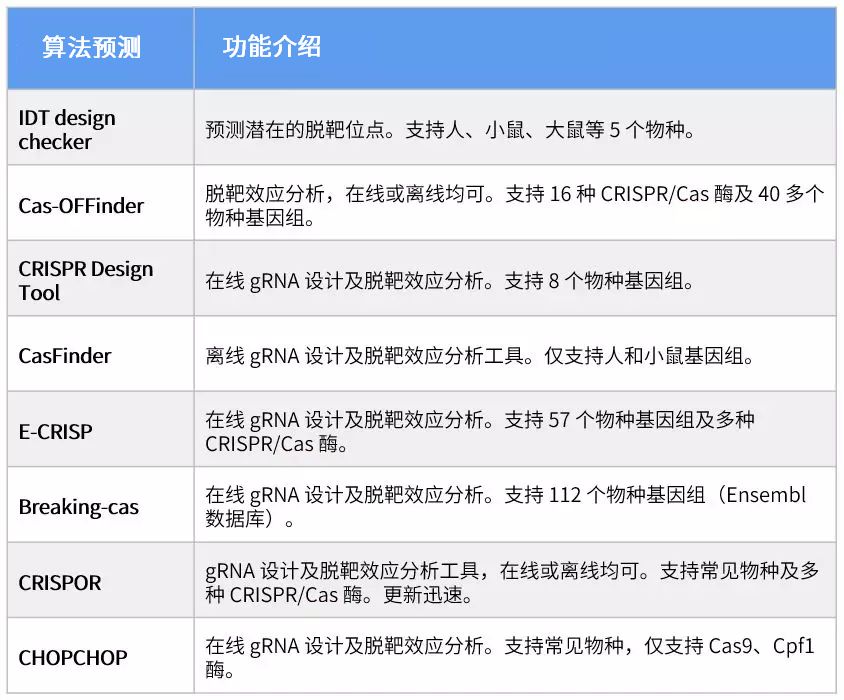
但是这些预测工具并未进行过生物信息学的系统性比较,每种算法都有自己的特点,且结果并不总是一致。对于基础研究用途,如制作基因突变的细胞系,算法预测验证可快速、简单、低成本的对数个位点进行脱靶分析。即使有误判或者漏掉脱靶位点,只要能排除对实验结果的影响,均属于可接受范围。
而在治疗药物开发、临床前研究等临床用途中,任何的潜在脱靶效应都可能对患者产生潜在的有害影响,必须进行全基因组范围的 CRISPR 脱靶效应检测。
02
体外基因组测序
Cas9 核酸酶和 gRNA 在体外可结合形成具有切割功能的核糖核蛋白复合物(Ribonucleoprotein complex,即RNP),RNP 与提取的基因组 DNA(gDNA,gnomicDNA)孵育即可进行切割,造成双链断裂。利用这一特性,可通过全基因组 DNA 测序来检测脱靶效应。目前常见的方法有以下 3 种:
表 2. 体外基因组测序分析脱靶效应
(点击查看大图)

这 3 种方法均可在全基因组范围内检测低至 0.1% 的脱靶位点,适合大规模筛选或用于临床分析。但考虑到 gDNA 与 RNP 直接反应,与细胞内真实发生的切割情况可能会有所区别。目前还需要更多与体内数据的对比,进一步研究完善。
03
基于细胞的基因组测序
对被 CRISPR 基因编辑过的细胞进行基因组测序,使得实验不局限于 RNP 系统,也可检测特定条件下的特定细胞。但是这种检测方式的灵敏度与实验方式、细胞类型等有关。常见的方法详见表 3:
表 3. 细胞基因组测序分析脱靶效应
(点击查看大图)

利用二代测序(NGS)从基因组水平上全局衡量脱靶效应是一个重要的议题。但是有两个直观的困难:一是测序成本,当样本数量和测序深度增加时,不同检测方法成本差异巨大,可能从几百元到几万元不等;二是生物信息学分析,目前暂时没有专业软件对测序结果进行脱靶效应分析。虽然有一些实验室已经编写了部分脚本代码分享,但并不具备通用性。
美国 Integrated DNA Technologies (IDT) 公司内部则采用基于细胞的基因组测序分析潜在脱靶位点,再使用自有专利的 rhPCR扩增子测序技术对脱靶位点进行进一步比较和研究。
解决方案
Cas9 酶本尊,是 CRISPR 脱靶效应的关键因素。IDT 提供的两种 Cas9 核酸酶:野生型 S.p. Cas9 和高保真型 S.p.HiFi Cas9,两者均有高效的编辑效率。前者为通用型,是大众首选;后者因其高保真性适用于对于脱靶效应十分敏感的场景。关于 IDT Alt- R® CRISPR-Cas 基因编辑系统具体介绍详见:是怎样的神操作 60 分钟进行 CRISPR 基因编辑。
IDT 对不同形式的野生型 Cas9 脱靶效应分析显示,RNP 模式(体外组装 tracrRNA:crRNA+Cas9)转染,虽然会引起剂量相关的脱靶,但仍远低于 crRNA:tracrRNA 转染进稳定表达 Cas9 的细胞系引起的脱靶效应(图 2.)[9]。其主要原因在于 RNP 在一定时间后被细胞降解,并不会长时间持续切割 DNA,因而脱靶效应也较低。
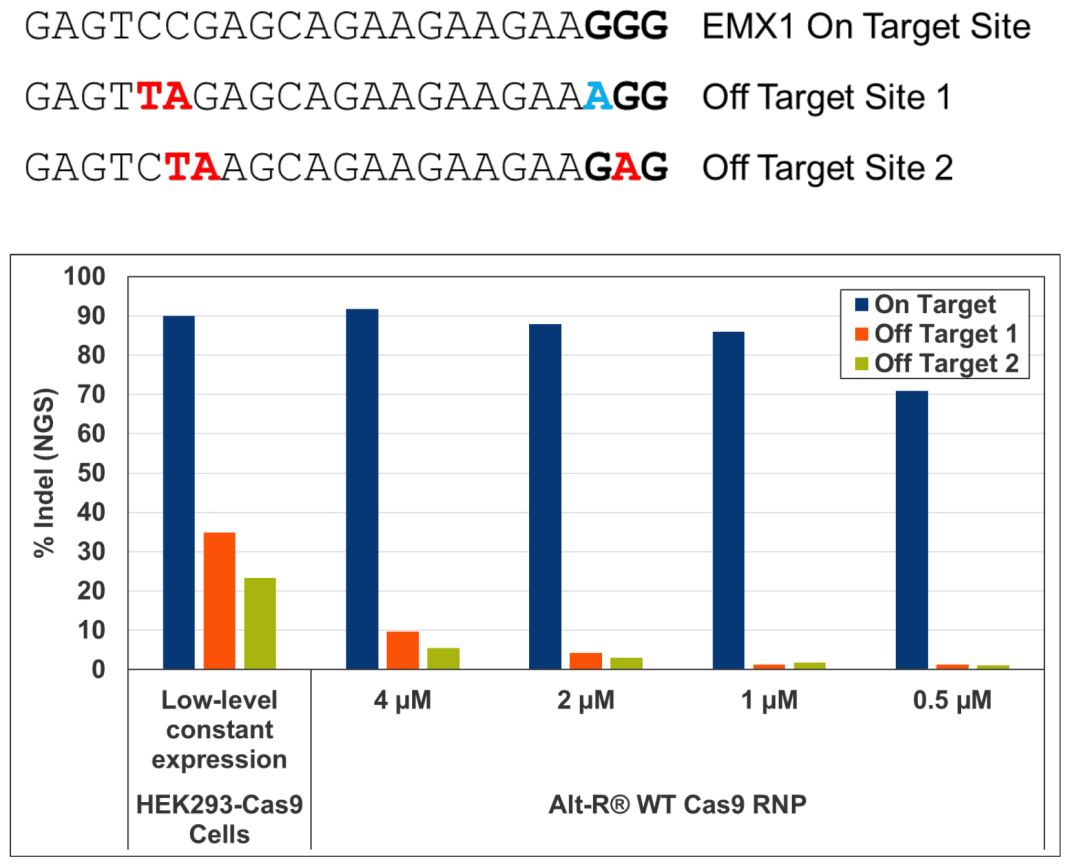
图 2. RNP 形式的野生型 Cas9 可降低脱靶效应。RNP 形式的脱靶效应远低于稳转 Cas9 的细胞系。IDT 针对 EMX1 靶向位点设计 Alt-R crRNA,与通用的 Alt-R tracrRNA 构成 gRNA,分别进行如下实验:1)电转至稳定表达 Cas9 的HEK-293 细胞系;2)与野生型 Cas9 形成 RNP,随后使用 Nucleofector 系统 (Lonza) 电转进 HEK-293 细胞系。之后对靶向位点和脱靶位点进行测序分析。
而 Alt-R® S.p. HiFi Cas9 的 RNP 形式或以其构建的稳转细胞系(图 3,第二列和第四列),脱靶比例都显著低于野生型(图 3,第一列和第三列)。HiFi Cas9 RNP 形式的脱靶效率则是最低的:在四个测试基因中都有高达 99% 的中靶率(图 3,第四列)[10]。
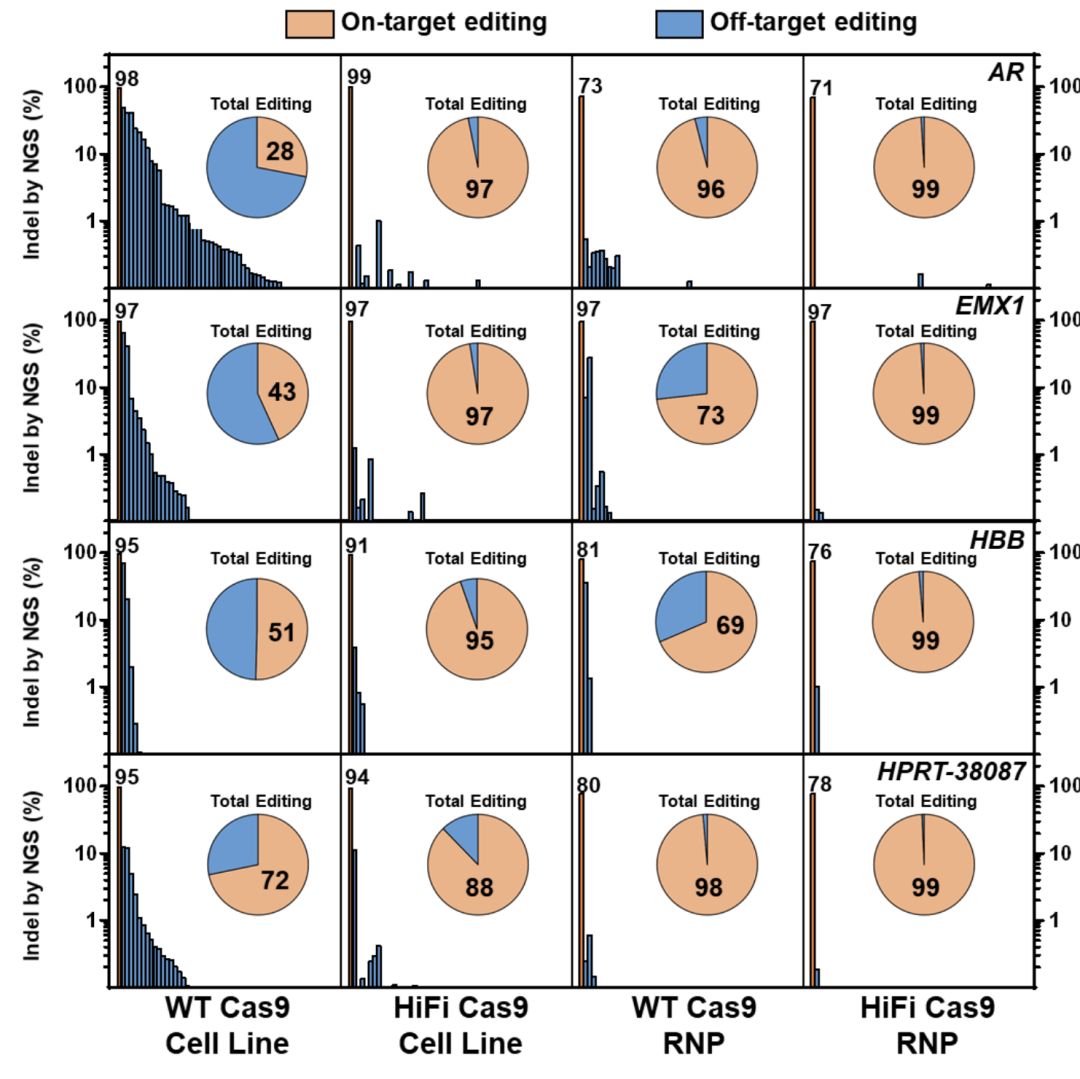
图3. AR、EMX1、HBB 和 HPRT 基因的靶向编辑 NGS 分析结果。不同的 gRNA 逆转染至稳定表达野生型和高保真型 Cas9 的细胞系中(第一列与第二列)和体外组装的 RNP 模式(第三列与第四列)。使用 GUIDE-Seq 检测所有的脱靶位点;之后利用 rhPrimer 扩增子测序方式分析每个位点的 indel 频率。
具体案例
镰刀型贫血症是由编码 β- 血红蛋白的基因 HBB 点突变而引起的遗传病。以往骨髓移植的疗法成功率低且难以解决排斥反应。CRISPR-Cas9 介导的精确同源重组(HDR)可修复病人自身造血干细胞的突变,但是基因编辑效率低,同时也存在脱靶的问题[11]。2018 年 4 月接收的一项最新研究里,来自美国斯坦福大学的研究团队则在 Alt-R® S.p. HiFi Cas9 的基础上优化,并建立了 RNP 结合同源重组修复 HBB 点突变的实验条件和方法。该方法拥有高效编辑效率的同时解决了脱靶的问题,为治愈镰刀型贫血症以及其他多种血液遗传病提供了坚实的基础[12]。
部分结果如图 4 所示,Alt-R® HiFi Cas9 在 CD34+ 的造血干祖细胞(HSPC)中实现对 HBB 基因稳定精确的靶向编辑,效率与野生型相对,同时脱靶效应又显著降低[13]。
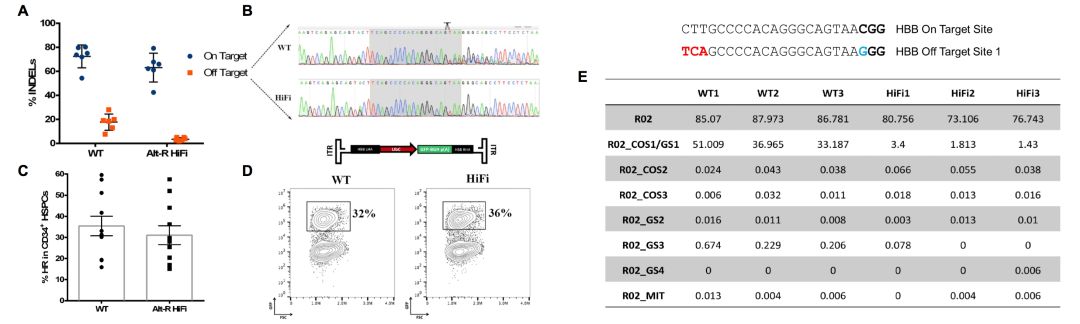
图 4. Alt-R HiFi Cas9 RNP 在 CD34+ 的 HSPC 中实现对 HBB 基因稳定精确的靶向编辑,同时脱靶效应显著降低。(A)野生型和高保真型 Cas9 分别以 1:2.5 的摩尔比与 HBB 特异的 gRNA 结合形成 RNP,电转至 CD34+HSPC。72~96 小时后提取基因组 DNA,PCR 扩增靶向区域和已知的脱靶区域。使用 TIDE 软件计算 INDEL 的频率。(B)Sanger 测序表明,Alt-R S.p. HiFi Cas9 酶(底部迹线)与 WT Cas9(顶部迹线)相比,脱靶效应显著减少。(C)将 CD34+HSPC 按上述方法电转后进行同源重组,通过流式细胞仪(FACS)分析插入 GFP 表达。可见 HiFi Cas9 酶与 WT Cas9 酶相比,同源重组率接近。(D)流式细胞仪分离 GFP 阳性的细胞考察同源定向修复(HDR)的频率。(E)对 HBB(R02)编辑效果进行 NGS 分析,GS1-4 为 GUIDE-Seq 的排名前四的脱靶位点,COS1-3、MIT 分别指的是 COSMID 和 MIT CRISPR 预测的脱靶位点。可见 HiFi Cas9 酶几乎无显著的脱靶位点[13]。
(转化医学网360zhyx.com)
 腾讯登录
腾讯登录
还没有人评论,赶快抢个沙发