剖析|你想要知道的数字PCR应用及前景
| 导读 | 一.PCR的发展历史PCR技术自问世以来,在遗传病、病原体、癌基因等分子诊断领域和法医鉴定等方面发挥了巨大作用。第一代PCR在进行扩增后通过凝胶电泳进行定性分析。随着生物分子荧光技术的发展,1992年实时荧光定量PCR(QuantitativeReal-timePCR,qPCR)应运而生。qPCR是一 |
一. PCR的发展历史
PCR技术自问世以来,在遗传病、病原体、癌基因等分子诊断领域和法医鉴定等方面发挥了巨大作用。第一代 PCR在进行扩增后通过凝胶电泳进行定性分析。
随着生物分子荧光技术的发展,1992年实时荧光定量PCR(Quantitative Real-time PCR, qPCR) 应运而生。qPCR是一种在DNA扩增反应中,以荧光化学物质荧光强度来测定每次聚合酶链式反应(PCR)循环后产物总量的方法。由于在PCR扩增的指数时期,模板的Ct值和该模板的起始拷贝数存在线性关系,所以Ct值也就成为定量的依据。基于荧光探针或染料的第二代 PCR 技术随后逐渐发展为检测核酸目标片段的主流分子诊断学技术。
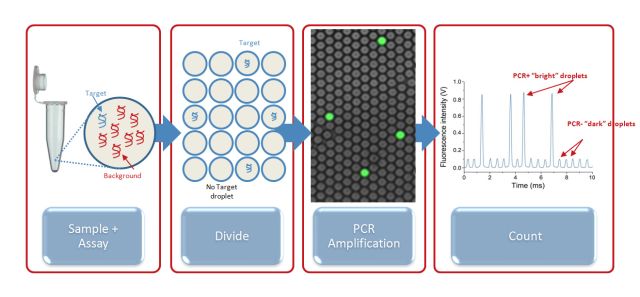
在实时定量 PCR(qPCR) 过程中,荧光信号随着扩增产物的积累而增强。qPCR 能够实时获得模板扩增的荧光值,然后根据DNA 模板在指数增长时期的 Ct 值与标准 DNA 的Ct值比较来计算初始模板的浓度。但是这种方法由于是大体积反应系统,非特异性的扩增增加了假阳性结果和背景信号,因此,最终无法获得绝对定量的结果。
随着MEMS工艺的不断成熟和微流控技术的不断发展,新一代PCR技术,数字PCR(digital PCR, dPCR) 也随之出现。digital PCR是一种新的绝对定量PCR,主要是对PCR反应物进行有限稀释,随后在不同的反应腔室里进行PCR扩增,最后根据泊松分布原理及阳性微滴的个数与比例得出靶分子的起始拷贝数或浓度的技术。
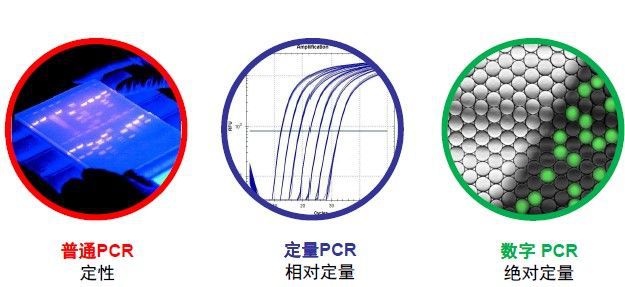
图1. PCR技术的发展历程
二. 数字微流控(dPCR)的原理
20 世纪末,Vogelstein 等提出数字PCR( digitalPCR,dPCR) 的概念,通过将一个样本分成几十到几万份,分配到不同的反应单元,每个单元包含一个或多个拷贝的目标分子( DNA 模板) ,在每个反应单元中分别对目标分子进行PCR 扩增,扩增结束后对各个反应单元的荧光信号进行统计学分析。
dPCR是一种核酸分子绝对定量技术。
dPCR一般包括两部分内容,即PCR扩增和荧光定量分析。
在PCR 扩增阶段,与传统技术不同,dPCR一般需要将样品稀释到单分子水平,并平均分配到几万个反应腔室里反应。这样子相当于变相的对靶基因进行富集。与此同时,由于对原样品的大幅度的稀释,使得PCR抑制剂浓度显著降低,这样dPCR对初始PCR反应物里抑制剂的要求显著低于qPCR。
不同于 qPCR对每个循环进行实时荧光测定的方法,dPCR 技术是在扩增结束后对每个反应单元的荧光信号进行采集,最后根据泊松分布原理及阳性微滴的个数与比例得出靶分子的起始拷贝数或浓度。
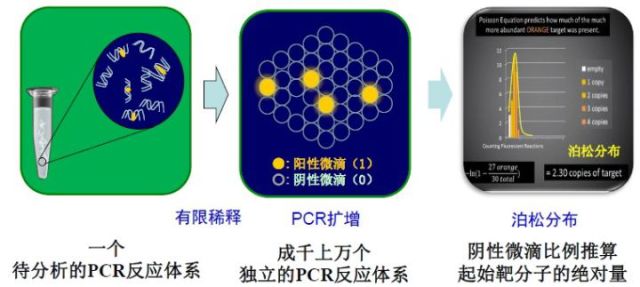
图2. dPCR基本原理
三. dPCR与qPCR的对比
相对于荧光定量PCR(qPCR)而言,dPCR具备以下优势:
1、 灵敏度可达单个核酸分子:检测限低至0.001%,原因在于dPCR可以实现靶标DNA/RNA的富集;

图3. dPCR可以实现痕量核酸的高灵敏检测
2、 无需标准品(标准曲线),即可对靶分子起始量进行绝对定量;
3、 特别适合基质复杂样品的检测:终点PCR检测,不依赖Ct值,不依赖扩增效率,能克服PCR抑制剂的影响,适合动血样、FFPE组织、粪便、尿液、痰液、水样、土壤、植物等复杂样品中DNA的绝对定量;
4、 能够有效区分浓度差异(变化)微小的样品:更好的准确度、精密度和重复性,可以用于精确测定靶基因的相对表达,基因拷贝数变异分析等。
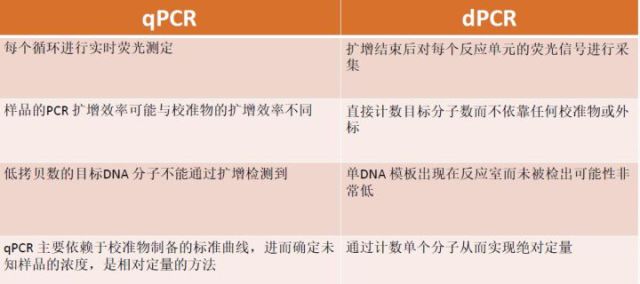
图4. qPCR和dPCR的对比
四.dPCR的多指标检测的实现
如同qPCR一样,dPCR中实现多指标的并行检测能显著降低检测成本,获取更丰富的检测信息。
不同于qPCR中的多腔室扩增与多荧光通道结合的并行PCR方式,在dPCR里,一个微反应腔室里一般只含一种靶基因。

图5. dPCR的多指标并行检测的实现方式
所以dPCR的多指标检测主要通过以下两种方式实现:
1. 多个荧光通道。使用多种荧光染料来标记不同的要检测的靶基因。
2. 单波长多强度。使用一种荧光染料,但是扩增结束时不同的靶基因对应的染料的荧光强度不一样。
五.dPCR的分类以及相关的产品
微流控芯片技术能够快速并准确地将样品流体分成若干个独立的单元, 从而进行多步平行反应,并且具有成本低、体积小和高通量等特点,是理想的数字PCR平台。按照其独立的单元的实现方式来细分,dPCR又分为基于液滴微流控(droplet microfluidics)的微滴数字PCR(droplet digital PCR, ddPCR)和基于芯片式微流控的微阵列芯片式PCR。
5.1微滴数字PCR(droplet digital PCR, ddPCR)
微滴数字PCR主要是将两种互不相溶的液体,以其中一种作为连续相(油),另一种作为分散相(水),在水/油两相表面张力和剪切共同作用下分散相以微小体积单元的形式存在于连续中,从而成液滴。这种液滴式的反应腔室具有体积小、样品间无扩散等优势。
在ddPCR中,利用微滴发生器可以一次生成数万乃至百万个纳升甚至皮升级别的单个油包水微滴,作为数字 PCR 的样品分散载体。这里,液滴中包裹了单拷贝DNA模板和PCR反应液,然后将液滴收集在PCR反应管中进行扩增。ØPCR反应结束后检测每个微滴的荧光信号。
目前Bio-Rad公司的QX100系统、QX200系统均为采用液滴技术的数字PCR系统。
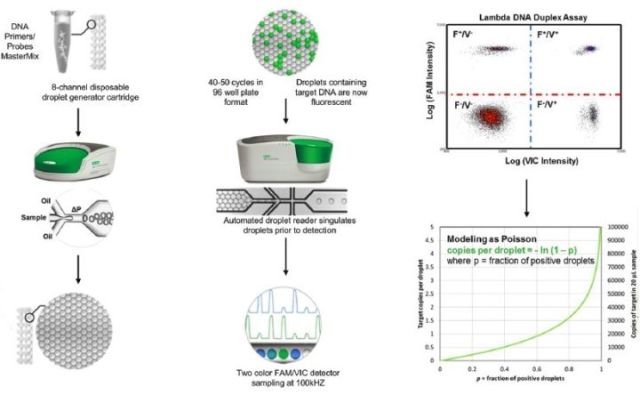
图6. Bio-Rad的droplet digital PCR系统
5.2 微阵列芯片式PCR
微阵列芯片式PCR主要通过芯片设计将纳升液体封闭在高通量的微池或微量通道中进行后续的PCR扩增及扩增后结果的荧光显微镜直接判读。
按芯片设计方式,微阵列芯片式PCR又可以分为阵列微池式芯片、滑片式芯片和集成微泵阀芯片:
1. 阵列微池式芯片上刻蚀有微池阵列,反应液由进样孔直接导入个反应微池;
2. 滑片式芯片是设计带有微流体通道和反应单元的玻璃芯片,上下两篇玻璃芯片间用油相密封,通过滑动芯片将样品溶液从液体通道引入反应单元,同时生成成百上千个微反应滴阵列;
3. 集成微泵阀式芯片是通过多层软刻蚀技术在聚二甲基硅氧烷(PDMS)芯片上加工交织的液体和气体通道结构,通过精确地控制微泵阀的开启和关闭,快速并准确地将流体分成若干个阵列的独立单元。
2010年,Life Technologies也推出了基于阵列微池式芯片的数字PCR产品线–OpenArray系统。它提供的TaqMan OpenArray 数字PCR试剂盒可在OpenArray平板上运行。平板可在现有的OpenArray 实时定量PCR系统以及新上市的QuantStudio™ 12K Flex仪器上运行。OpenArray 实时定量PCR系统能够同时运行3块OpenArray平板。

图7. TaqMan® OpenArray® Digital PCR系统
Fluidigm公司于2006年底推出了基于集成流体通路(IFC)芯片的Bio-Mark™ 高通量基因剖析系统。
其创新在于集成液体通路技术:应用集成电路制造工艺(光刻)在硅片或石英玻璃上刻上许多微管和微腔体,经过不同的控制阀门控制溶液在其中的活动来完成生物样品的分液、混合、PCR扩增。

图8. Bio -Mark™ IFC芯片
六.dPCR的应用前景
6.1 痕量核酸检测

图 9. 痕量核酸检测的具体分类
dPCR尤其适合:对灵敏度要求非常高的核酸痕量分析研究;基质复杂样品中的核酸准确定量(如组织、体液、排泄物等样品)。

图10. 痕量核酸检测相关方法比较
6.1.1 癌症标志物稀有突变检测
在一份给定的样本中,相比于野生型DNA,癌症相关的突变序列比例较低,经常无法检测。而凭借其超高灵敏度,dPCR系统可以很容易地定量分析低至0.001%-0.0001%的突变频率。之前用任何方法都无法实现准确稳定检测的样本,现在可以使用dPCR轻松进行定量分析。
是否能够开发出有效的靶向治疗药物,与突变基因的检测密切相关。其中(1)携带药物作用突变位点的癌细胞百分比;(2)突变的特定等位基因是否可以被准确检测到,这两点都直接影响到靶向治疗是否有效。很显然,在肿瘤均质细胞内检测单一突变相对简单,但是如果在异质组织内,在仅存有少量突变存在的情况下,如何准确检测出突变/稀有变异,或者针对于同一种癌症中多种亚克隆的准确鉴定,对个性化医疗的发展是一个巨大的挑战。
案列1. 实现肺癌相关的表皮生长因子受体(EGFR)中T790M突变的检测,可以更好地评估抗酪氨酸激酶抑制剂的治疗。然而,由于其他技术检测灵敏度有限,无法在高背景EGFR野生型中可靠地检测到T790M突变。研究人员使用dPCR技术开发出精确度很高的检测方法,以准确定量T790M的浓度[1]。

图11. ddPCR可以用EGFR突变的肺癌患者的血浆中游离DNA(cfDNA)为样本,检测与EGFR敏感性和药物抗性相关的突变
6.1.2 致病微生物检测
精确的病毒载量分析对于阐释疾病病程,后续治疗及疗效评估是至关重要的。病毒载量的波动,即使只下降了非常低的水平,意义却是显著的。在对病原体的研究中,能否实现准确而可靠的病毒DNA或RNA定量分析,关系到实验的成败。
案列2. 研究人员比较了 qPCR 和 dPCR 方法检测临床样本中残留的人类免疫缺陷病毒(HIV),其中包括功能性治愈的HIV感染婴儿。研究人员发现,在检测百万个细胞内的 HIV DNA拷贝数时,相比于 qPCR,dPCR 的灵敏度提升了5倍;在检测病毒长末端重复序列时,dPCR比qPCR灵敏提升了20倍[2]。
案列3. 研究人员探索HBV血清学和ddPCR检测结果与肝细胞癌(HCC)临床分期和病症之间的关系中,ddPCR成功的克服了real-time PCR(qPCR)在检测FFPE处理的肝癌患者组织样品中HBV DNA时,遇到的准确度、灵敏度和重复性不足的缺点。进而得出以下结论: 1、ddPCR的灵敏度和准确度比real-time PCR更高——131个待测样品中HBV DNA浓度范围为1.1~175.5 copies/ul; 2、即使是在24个血清学检测呈阴性的样本中也检测到了痕量的HBV DNA; 3、样品中HBV DNA的含量与肝细胞癌(HCC)组织的临床分期一致; 4、ddPCR技术可以用于肝癌的早期无创诊断,病程监控,肝移植、化疗或免疫抑制的疗效评估[3]。
6.2 与新一代测序(NGS)的无缝对接
相对于NGS, dPCR可以富集待测序样品中的靶基因; dPCR还可以验证 / 精确定量测序结果。
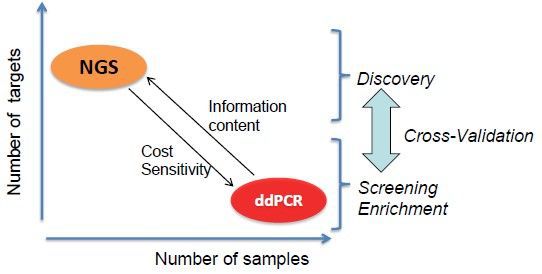
图12. ddPCR和NGS的优势互补
案列4. 由于人间充质干细胞(MSC)在人体内的含量极低,所以目前应用于临床治疗的MSCs都来源于体外培养。研究人员分别对体外培养p1,p8和p13阶段(passage)的来源于人骨髓MSC采用全基因组测序(WGS)。WGS结果显示在p1和p8阶段的培养细胞中没有拷贝数变异(CNV)和非常低频的单核苷酸突变(SNV),但是p13阶段的SNCs数目显著增加,达到677个。采用ddPCR对WGS发现的8个非同义突变进行了确认,结果发现在未经培养的单核细胞内就含有极低频对应的突变(0.01%),在p1和p8阶段培养的MSC内其突变比例依然处于极低比例状态(0.1-1%),但是在p13培养的阶段对应突变的比例显著地上升到(17-36%)[4]。
6.3 基因表达分析
实时荧光定量PCR 常用于检测基因表达差异,但该方法一般只能检测出两倍或者更大的差异。而一些研究则需要检测低于两倍的表达变化。dPCR 的检测精度可达±10%甚至更高,能够分辨更小的差异。
dPCR尤其适合:基因相对表达变化差异较小(<2倍)的研究;低丰度基因或单细胞的表达分析,以及RNA编辑、等位基因差异表达等研究。
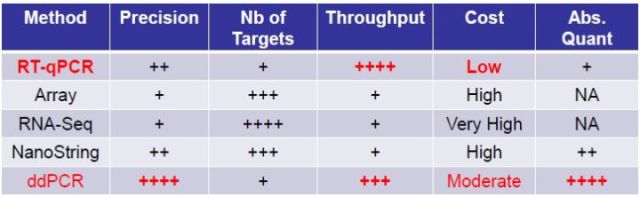
图13. 基因表达分析相关方法比较
6.4 拷贝数变异分析

图14. 拷贝数变异
拷贝数变异 (CNV) 分析的目的是确认目的序列的拷贝数是否偏离野生型序列,以及偏差多少。拷贝数变异与疾病(癌症)、代谢途径、生物种进化等关系密切。临床上,CNV可为具体治疗方案的制定与修正提供参考;农业及畜牧业,CNV可作为遗传育种的遗传标记。
传统实时荧光定量PCR平台提供了足够的分辨率,可以鉴别低拷贝数 (如0至5的拷贝数);但更高的拷贝数需要更精确的测量,以确定确切的拷贝数。dPCR尤其适用于更高拷贝数分析,检测精度超过±10%。
参考文献:
1. Oxnard G R, Paweletz C P, Kuang Y, et al. Noninvasive detection of response and resistance in EGFR-mutant lung cancer using quantitative next-generation genotyping of cell-free plasma DNA[J]. Clinical cancer research, 2014, 20(6): 1698-1705.
2. Persaud D, Gay H, Ziemniak C, et al. Absence of detectable HIV-1 viremia after treatment cessation in an infant[J]. New England Journal of Medicine, 2013, 369(19): 1828-1835.
3. Huang J T, Liu Y J, Wang J, et al. Next generation digital PCR measurement of hepatitis B virus copy number in formalin-fixed paraffin-embedded hepatocellular carcinoma tissue[J]. Clinical chemistry, 2015, 61(1): 290-296.
4. Cai J, Miao X, Li Y, et al. Whole-genome sequencing identifies genetic variances in culture-expanded human mesenchymal stem cells[J]. Stem cell reports, 2014, 3(2): 227-233.
(转化医学网360zhyx.com)
 腾讯登录
腾讯登录
还没有人评论,赶快抢个沙发