CRISPR技术再升级——单碱基的精准编辑
| 导读 | 美国哈佛大学David R. Liu课题组创新性的开发了基于CRISPR/Cas9的单碱基编辑器(base editor, BE),有效地解决了精准修复致病基因点突变难题。 |
CRISPR/Cas9基因组编辑系统来源于简单的细菌免疫系统组分,经过改造后可在真核细胞中实现高度灵活且特异的基因组编辑。该系统是单RNA(single-guide RNA, sgRNA) 介导的核酸酶系统,通过靶点特异的CRISPR RNA (crRNA)序列与靶序列进行碱基配对从而引导Cas9蛋白至特定的切割位点进行切割,然后通过经典的非同源性末端接合 (NHEJ) 或同源重组 (HDR) 对断裂的DNA进行修复。然而,NHEJ修复会造成核酸的插入或者缺失(Indel), 产生显性负效应突变或者新抗原表位的风险。虽然HDR修复精确度高,然而HDR修复效率极低且局限于有丝分裂细胞,限制了HDR修复的应用。因此,精准修复致病基因点突变依然是一大难题。
美国哈佛大学David R. Liu课题组发表在《Nature》的” Programmable editing of a target base in genomic DNA without double-stranded DNA cleavage”论文创新性的开发了基于CRISPR/Cas9的单碱基编辑器(base editor, BE),有效地解决了上述问题。
该研究组将胞嘧啶(C)转变为尿嘧啶(U)的大鼠胞嘧啶核苷脱氨酶rAPOBEC1通过linker序列XTEN连接到催化失活Cas9(catalytically dead Cas9, dCas9)N端,构建出了第一代单碱基编辑器base editor (BE1)(rAPOBEC1–XTEN–dCas9)(图一)。
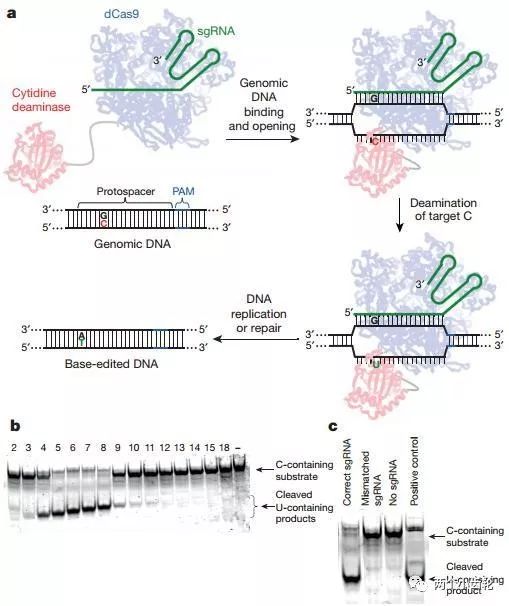
图一
运用BE1进行体外实验表明目标C碱基5端是G碱基时编辑效率显著降低,BEI编辑效率遵循TC ≥ CC ≥ AC > GC (C为目标碱基)原则,此外目标C碱基位于sgRNA的第7位或者附近时编辑效率最高(图二)。体外细胞实验研究表明,运用BE1可以使C→T的编辑效率达到0.8% - 7.7%(图三)。

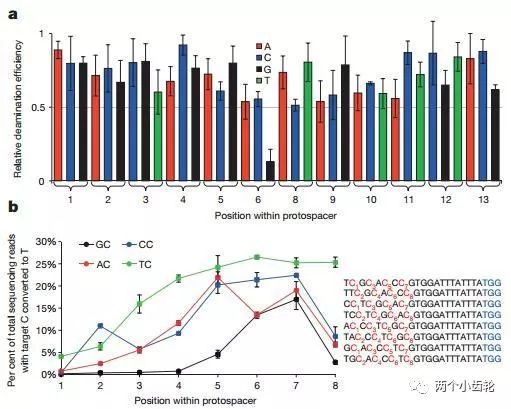
图二
鉴于BE1的单碱基编辑效率较低,研究者们认为有可能是BE1编辑使CG碱基对转换成UG碱基对,然而正常细胞会将UG识别为错配碱基对,从而启动体内尿嘧啶DNA糖基化酶(Uracil DNA glycosylase, UDG)识别U并将其切除,进而将其重新修复为CG。因此在BE1的基础上,研究者们又融合了尿嘧啶DNA糖基化酶抑制剂(UGI),构建出了第二代单碱基编辑器BE2(rAPOBEC1–XTEN–dCas9-UGI)。实验结果表明BE2编辑效率是BE1的3倍(图三)。
BE2编辑过程中只改变了CG碱基对中的C碱,因此其理论上C→T的最大转换效率只能达到50%。为了进一步提高编辑效率,研究者们部分恢复BE2编辑器中Cas9的切割活性(dCas9-(A840H)),构建出了第三代单碱基编辑器BE3(rAPOBEC–XTEN–dCas9(A840H)–UGI)。BE3中的Cas9可以切割包含G碱基的非编辑DNA单链,断裂后的非编辑链(G所在链)会以编辑链(T所在链)为修复模板进行修复,因此理论上C→T的转换效率可以达到100%,即CG可以完全转换为TA。研究结果表明BE3编辑效率是BE1的2-6倍(图三)。
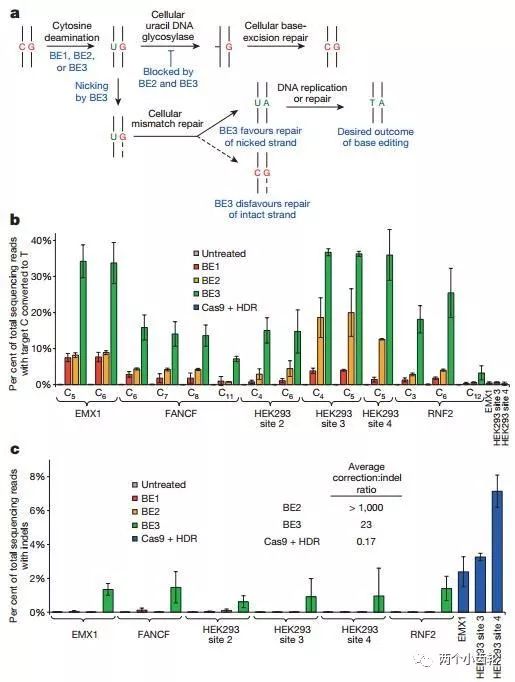
图三
最后,研究者们运用BE3对致病突变修复进行了研究。在体外细胞系的试验发现BE3对阿尔茨海默病致病基因APOE4的改造效率可以高达74.9%,对癌症相关致病基因TP53的改造效率为7.6%,而indel率维持在较低水平(4.6%及0.7%)(图四)。
BE3单碱基编辑技术的出现为真正意义上的单碱基(T→C, A→G)致病突变基因的精准修复提供可能。
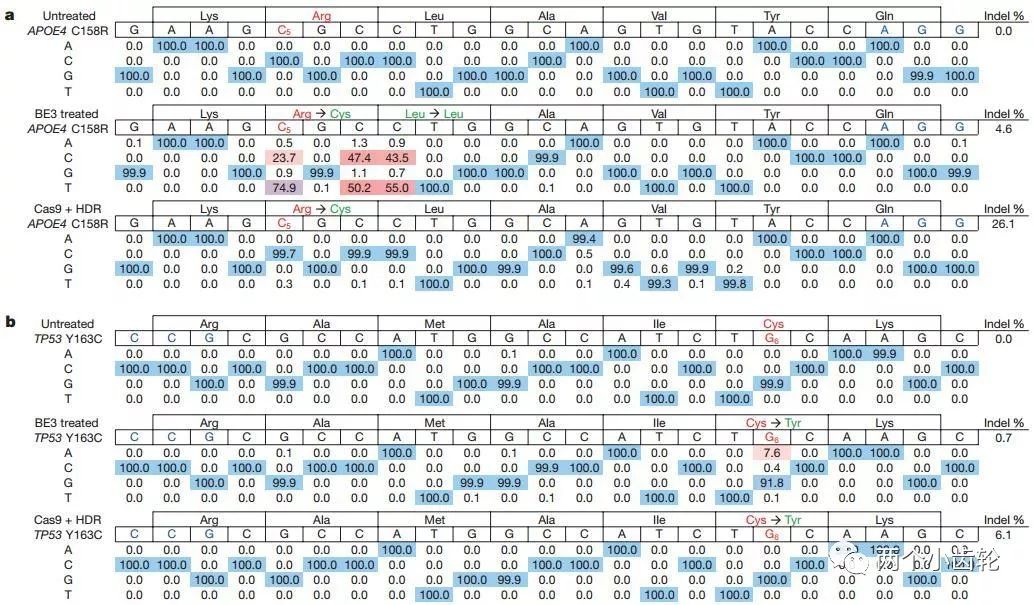
图四
(转化医学网360zhyx.com)
 腾讯登录
腾讯登录
还没有人评论,赶快抢个沙发