【研究前沿】同济大学张赫团队发表关于染色质拓扑相关结构域调控基因转录新模式的最新研究
| 导读 | 2024年5月8日,同济大学生命科学与技术学院张赫团队在《基因组蛋白质组与生物信息学报》(Genomics Proteomics Bioinformatics)发表了题为“Inter3D: Capture of TAD Reorganization Endows Variant Patterns of Gene Transcription”的最新研究成果 |
2024年5月8日,同济大学生命科学与技术学院张赫团队在《基因组蛋白质组与生物信息学报》(Genomics Proteomics Bioinformatics)发表了题为“Inter3D: Capture of TAD Reorganization Endows Variant Patterns of Gene Transcription”的最新研究成果,首次建立了一种名为Inter3D的新方法,利用该方法特异性地识别TAD重构介导的功能染色质环,揭示基因转录调控新模式。
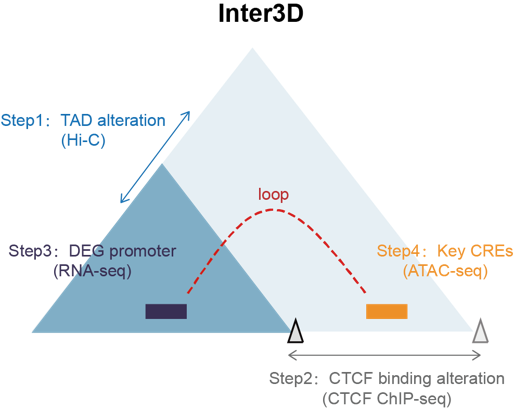
Inter3D分析方法用于分析顺式转录调控元件(cis regulatory element,CRE)与基因启动子间的相互作用。TAD重构存在两种不同机制,即Intra-TAD(Mechanism 1,CTCF促进启动子–CRE相互作用)或Inter-TAD(Mechanism 2,CTCF阻碍启动子–CRE相互作用),在调控基因转录方面发挥着关键作用。Inter3D首先使用Hi-C和CTCF ChIP-seq数据集来识别CTCF介导的TAD边界变化,并结合RNA-seq数据集识别出由CTCF结合变化和TAD重构调控的显著差异表达基因(differentially expressed genes,DEGs),再通过ATAC-seq数据集的叠加,在启动子远端的峰值中识别候选增强子,排除仅受三维结构而非增强子调控的假阳性靶基因。通过Inter3D方法,该研究成功计算出基于Inter-TAD(Mechanism 1)的8个癌基因和28个抑制基因,以及基于Intra-TAD(Mechanism 2)的11个癌基因和26个抑制基因。
为了进一步检测Inter3D方法的准确性,该研究挑选了抑癌基因MYL12B和促癌基因CYP27B1。40 Kb分辨率结果显示,相较于正常细胞ARPE19,在肿瘤细胞WERI-RB1中,18号染色体上的MYL12B基因座处存在一个新生TAD(neo-TAD)边界,发生TAD分离现象,导致MYL12B启动子(P1)与其下游增强子(E1)之间染色质环特异性丢失,引起WERI-RB1细胞中MYL12B转录水平显著降低。

此外,与ARPE19细胞相比,WERI-RB1细胞12号染色体上的促癌基因CYP27B1位点发生TAD融合现象,使得CYP27B1的启动子(P2)和增强子(E2)共存于同一个TAD中,形成特异性染色质环,导致肿瘤细胞中CYP27B1转录上调。

同济大学丁天宜、傅沙镠与张晓宇为该文共同第一作者,同济大学刘琦、张赫为该文共同通讯作者。同时,作为同济大学对口支援井冈山大学的联合学术成果,该工作获得了同济大学生命科学与技术学院、同济大学附属东方医院、江西省器官发育与表观遗传重点实验室、井冈山大学附属医院临床医学研究中心、井冈山大学医学部和生命科学学院的大力支持,该工作还受到了国家重点研发计划、国家自然科学基金委和上海市科委等项目的资助。
论文链接:https://academic.oup.com/gpb/advance-article/doi/10.1093/gpbjnl/qzae034/7667293

-
 游客2024-06-04 21:47:24e
游客2024-06-04 21:47:24e
 正在加载
正在加载 腾讯登录
腾讯登录