【Nature子刊】仁济医院王博石团队揭示转座元件助力肝癌研究
| 导读 | 本研究旨在探讨转座元件相关分子,在肝癌细胞中的功能和临床意义,重点关注肝脏特异性,及TE (liver-TEs) 调节邻近基因表达的机制。 |
2024年7月4日, 上海交通大学附属仁济医院上海肿瘤研究所王博石团队在期刊《Nature Communications》上发表了题为“Transposable elements-mediated recruitment of KDM1A epigenetically silences HNF4A expression to promote hepatocellular carcinoma”的研究论文。本研究揭示了促进肝癌生长的肝脏-TE/KDM1A/HNF4A调控轴,并强调KDM1A是一个有前途的治疗靶点。团队的研究结果,提供了对肝癌进展背后的转座元件相关分子机制的见解。

https://www.nature.com/articles/s41467-024-49926-2
研究背景
01
转座元件或转座子,是存在于大多数真核生物基因组中的移动DNA单元。虽然它曾经被认为,是没有功能意义的“垃圾DNA”;但最近的研究表明,TE提供了连接转录调控网络的调控序列。虽然宿主生物体通常会沉默TE,但TE衍生的顺式调节元件(CRE)的残余,可以持续存在,并适应控制宿主基因的转录。
TE以错综复杂的方式与癌症相互作用,为癌症的发展和进展机制,提供了新的渠道。TE可以作为启动子发挥作用,驱动癌基因表达和肿瘤发生,从而促进肿瘤的发生和维持。它们可以建立涉及KRAB锌指蛋白家族的调节回路,来抑制肿瘤。此外,TE可以与表观遗传事件(如DNA甲基化和组蛋白修饰)相互作用,以调节基因表达模式,并促进肿瘤生长。TE参与肿瘤表观遗传调控,代表了TE沉默,以维持基因组稳定性的结果。抑制TE的主要机制,涉及抑制性组蛋白修饰的选择性沉积。肿瘤细胞采用这种策略来促进免疫逃逸,其他表观遗传调节因子,如人类沉默枢纽(HUSH)复合物和赖氨酸去甲基化酶1A(KDM1A),采用类似的TE依赖性机制,来影响肿瘤发展。
KDM1A是一种公认的致癌基因,在多种癌症中,表现出高表达,并与癌症患者的不良预后相关。在肝癌中,KDM1A通过调节FKBP8/Bcl2或LINC01134/SP1/p62等信号,促进癌症生长和耐药性。KDM1A通常作为复合物的一部分发挥作用,例如CoREST复合物,它选择性地去除H3K4me1和H3K4me2甲基化修饰,以抑制靶基因的转录。或者,在具有激素受体的复合物中,KDM1A特异性调节H3K9me2和H3K9me3甲基化修饰。KDM1A对组蛋白底物的选择性,取决于复合物的组成。HNF4A是核受体超家族的成员,是肝癌发展初始阶段的关键调节因子。在正常生理条件下,HNF4A在肝组织中表现出高表达,在维持肝细胞的分化和功能中起关键作用。然而,HNF4A在肝癌中的表达下调,并显示出肿瘤抑制特征。
在本研究中,团队展示了KDM1A表观遗传,抑制HNF4A基因附近的转座元件,导致抑制HNF4A表达的机制。这项工作的重点,是提供CRE并募集KDM1A,以沉默附近基因的肝脏特异性可及TE(肝脏-TE)。此外,团队发现了肝脏TE、KDM1A和肝脏TE相关基因(HNF4A)协同控制肝癌细胞增殖的信号轴。这些发现,阐明了转座元件介导的分子事件的功能和临床意义,有望为肝癌细胞的新治疗策略铺平道路。
研究进展
02
KDM1A富含肝脏TE,是HCC细胞生长所必需的
为了研究肝脏TEs的表观遗传特征,团队使用RemapEnrich算法,分析了肝脏TEs中富集的组蛋白标记。结果显示,肝脏TEs富集于H3K4me1、H3K4me2和H3K27ac等活性组蛋白标记中。此外,团队对肝脏-TEs中CpG甲基化水平的调查表明,它们与非肝-TEs中的CpG甲基化水平相当,DNA甲基化不会显著影响肝脏TE活性。
为了阐明肝脏TE调控基因表达的分子机制,团队对肝脏TEs上的转录调节因子(TR)富集,进行了RemapEnrich分析。研究结果显示,组蛋白去甲基化酶KDM1A,在肝脏-TE上大量富集。在KDM1A直接靶向的区域中,H3K4me1在KDM1A敲低后表现出上调,而H3K9me2没有显著变化。值得注意的是,正常肝细胞中KDM1A的减少,并未导致缺乏KDM1A结合峰的肝TE的H3K4me1修饰升高。这些结果表明,KDM1A可能直接调节H3K4me1。
为了进一步证实我们的发现,团队用几种KDM1A小分子抑制剂处理HepG2细胞,并观察到与KDM1A敲低细胞相似的H3K4me1变化。团队发现,在总共1,923个肝脏-TE相关基因中,有1,202个基因被KDM1A靶向,约占队列的62.5%。RNA-seq和GSEA检测表明,敲低KDM1A,导致肝脏TE相关基因的整体表达升高。说明KDM1A在调控这组基因中具有全局抑制作用,并协调对肝脏TE的抑制作用。
集落形成试验表明,KDM1A下调后,可显著抑制肝癌细胞生长。而KDM1A过度表达,则促进细胞生长。为了评估与其他组蛋白甲基化调节剂相比,KDM1A在肝癌细胞生长中的相对重要性,团队对DepMap数据库中,RNAi筛选结果得出的平均依赖性评分,进行了排名。结果表明,肝癌细胞对KDM1A具有高度依赖性。此外,用小分子抑制剂抑制KDM1A的药理学,可显著减弱肝癌细胞的生长。SP2509治疗后,肿瘤生长明显延迟,而体重没有变化。HNF4A-liver-TEs和KDM1A敲低的耗竭,对肿瘤细胞生长具有显著的抑制作用。此外,利用HCC-PDO模型,团队研究了HNF4A-liver-TEs耗竭和KDM1A抑制,对HCC细胞3D生长的影响,揭示了KDM1A抑制对3D生长的显著抑制。综上所述,上述研究结果表明,靶向KDM1A,可能是一种有前途的调节肝脏TE活性的方法,也是治疗HCC的潜在策略。

抑制KDM1A调节肝脏TEs,并阻碍HCC细胞生长。
KDM1A 高表达与肝癌细胞预后不良相关
团队使用90份肝癌细胞组织样本,对KDM1A进行了免疫组织化学染色和生存分析,发现KDM1A的高表达,表明肝癌细胞预后不良。此外,使用TCGA、ICGC和GSE14520数据集的生存分析表明,高KDM1A表达组患者的总生存期(OS)和无进展生存期(PFS),明显短于低表达组。团队对用于KDM1A分析的同一组组织样本,进行了免疫组织化学染色,以检测ZMYM3和HNF4A的表达。随后的相关性分析显示,KDM1A和ZMYM3与HCC样品中,HNF4A的表达呈负相关。此外,团队分析了TCGA和ICGC肝癌细胞表达谱数据集中,KDM1A表达水平、ZMYM3表达水平、肝TE相关基因表达谱和HNF4A下游基因表达谱的临床相关性。结果表明,KDM1A和ZMYM3的表达水平,与肝脏TE相关基因和HNF4A下游基因的表达谱,呈显著负相关。
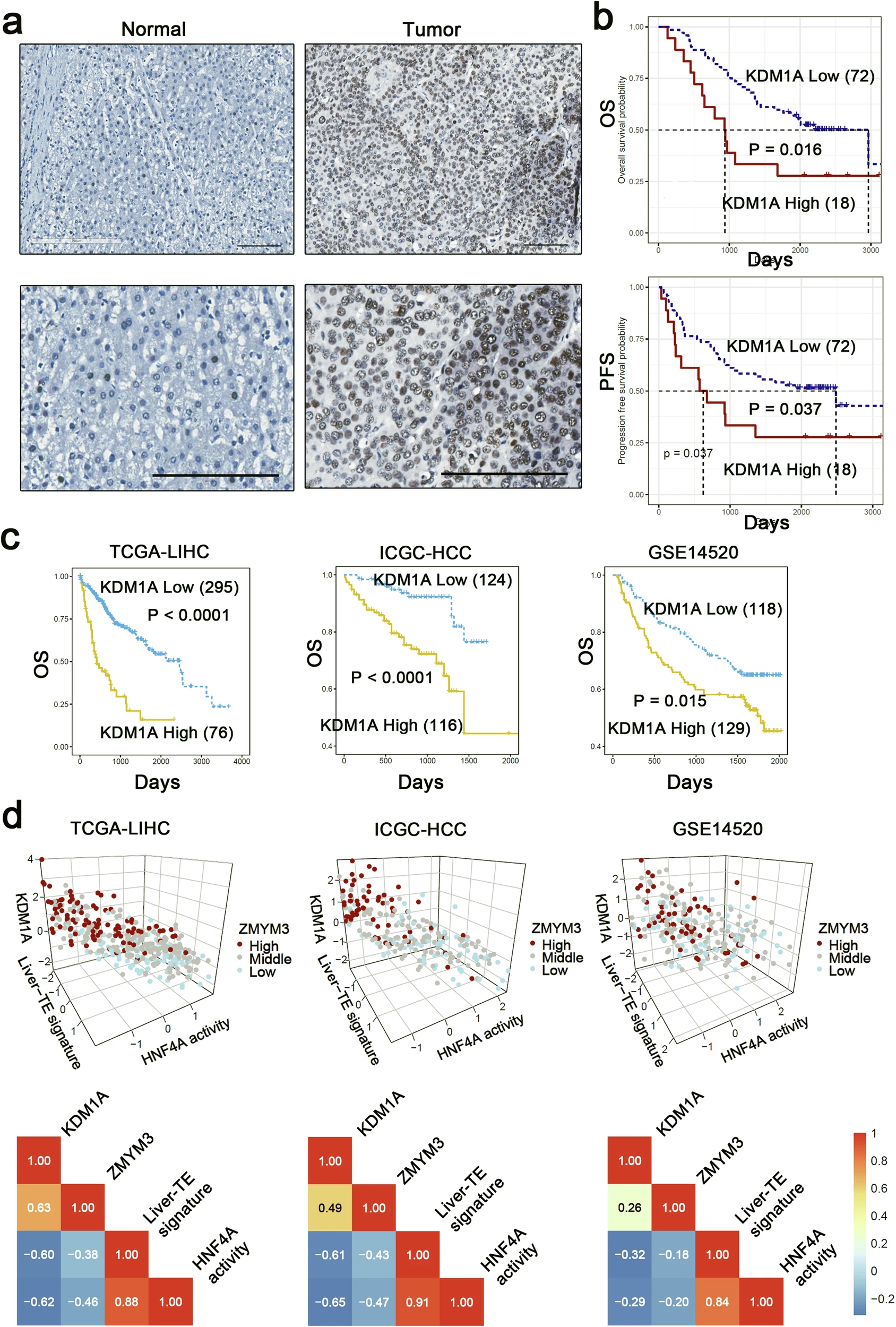
KDM1A在肝癌细胞中过度表达,预后不良。
研究结论
03
在本研究中,团队揭示了肝脏TE,在肝癌中的功能作用和重要性。团队鉴定了富含组蛋白标记、表观遗传调节因子的肝脏TE,并与一组与不良预后负相关的肝组织特异性基因相邻。肝TE相关基因在肝组织和肝癌中,均高度表达。肝脏TE相关基因,包括已知的肝癌抑制基因,HNF4A34。具体来说,团队重点研究了肝脏TE对HNF4A基因表达的调控作用。通过使用CRISPR/Cas9 技术,团队选择性地去除了位于肝癌细胞中HNF4A基因转录调控区(HNF4A-liver-TEs)内的肝脏TE。值得注意的是,这种肝脏-TE的缺失,导致HNF4A表达和肿瘤细胞生长抑制的显著上调,为这种类型的DNA元件对HNF4A的抑制功能,提供了令人信服的证据。
团队的研究结果表明,组蛋白标记物H3K4me1及其去甲基化酶KDM1A,在肝脏TE处富集,说明该区域染色质状态的复杂调节。H3K4me1通常与增强子区域相关,其存在是活性增强子的标志。然而,KDM1A是一种去甲基化酶,可去除组蛋白中的H3K4me1标记,从而导致基因抑制。H3K4me1和KDM1A在肝脏TE上的共富集,似乎是矛盾的,但团队为这一观察结果,提出了几种可能的解释。
一种可能的解释是,KDM1调节肝脏TE位点的H3K4me1水平,以微调其转录调节活性。KDM1A 已被证明,可作为转录共抑制因子发挥作用,并可能与其他染色质调节因子协同作用,以沉默基因表达。在这种情况下,KDM1A在肝脏-TE中的存在,可以通过调节H3K4me1的水平,来帮助防止附近基因的异常激活。另一种可能性是,肝脏-TE代表了活性和非活性增强子之间的过渡状态,其中H3K4me1和KDM1A共存。在这种情况下,H3K4me1和KDM1A水平之间的平衡,可能决定TE衍生的CRE,是活跃的还是抑制的。低水平的KDM1A,可能允许H3K4me1在TE 衍生的CRE周围持续存在,并维持活性状态;而高水平的KDM1A,可能导致H3K4me1去除和随后的沉默。总体而言,H3K4me1和KDM1A在肝脏TE上的共富集,表明该区域的反式激活活性,具有复杂的调控作用。这与肝癌中KDM1A的高表达,和肝癌中肝TE相关基因的低表达一致。这为肝癌中KDM1A与肝脏TE相关基因表达之间的内在联系,提供了机制上的见解。
CRISPRa在HNF4A-liver-TE背景下的应用,也揭示了KDM1A的显著调控作用。虽然CRISPRa技术通常用于诱导靶向基因表达,将会导致转录激活;但团队的研究结果,呈现出一种微妙的情况。团队观察到HNF4A-liver-TE的CRISPRa靶向,导致抑制染色质状态。对这一观察结果的合理解释,是CRIPSRa诱导的TE的瞬时暴露,随后募集KDM1A。因此,尽管CRISPRa预期激活,但 KDM1A的募集导致,HNF4A 表达的抑制。这些结果突出了,基于CRISPRa的操作,与靶区环境之间错综复杂的相互作用。有必要进一步研究CRISPRa的这些非典型效应背后的确切机制,因为它们可能为开发更精细的基因操作策略,提供有价值的见解。
团队的研究结果,强调了KDM1A通过涉及HNF4A的两种机制,在调节肝癌细胞生长中的关键作用。首先,KDM1A促进去甲基化,并限制肝脏TE的可及性,导致相关基因的抑制。团队的研究表明,KDM1A通过与HNF4A TRR 内的肝脏TE结合,并抑制其表达,直接调节HNF4A。说明KDM1A参与抑制HNF4A 表达。其次,KDM1A与HNF4A复合物相互作用,在表观遗传学上沉默下游基因,影响HNF4A的表达水平和转录活性。HNF4A是肝脏TEs中富集的转录调节因子中的一个重要因子,其靶基因高度富集于肝脏TE相关基因中。这种相互作用和表观遗传调控,有助于KDM1A抑制肝脏TE相关基因。团队的研究结果,揭示了KDM1A和HNF4A对肝癌生长的调控机制,突出了它们在肝脏TE可及性和转录活性中的重要作用。肝脏-TE/KDM1A/HNF4A调节环路,说明了这些因素在肝癌发展和进展中,错综复杂的相互作用。
团队的研究结果,表明了ZMYM3的功能意义,突出了其增强KDM1A的DNA结合特性,并将其调节功能引导至特定靶位点的能力。这种作用机制,与蛋白质复合物在调节基因表达中的更广泛作用一致。其中复合物的特定组成,决定了其与特定DNA序列结合,并调节相关酶活性的能力。
KDM1A的高表达,与肝细胞癌患者的不良预后有关,表明其作为预后生物标志物的潜力。KDM1A和ZMYM3的表达水平,与肝TE相关基因和HNF4A下游基因的表达谱呈负相关,强调了分子水平发现的临床相关性。敲低KDM1A在体外和体内,均显著抑制肝癌细胞生长,突出了KDM1A在HCC细胞生长中的重要作用。此外,使用靶向KDM1A的小分子抑制剂,可显著抑制肝癌细胞的生长,表明KDM1A是HCC治疗的有希望的靶点。使用KDM1A小分子抑制剂,可能是HCC治疗的可行策略,需要进一步研究,以探索KDM1A作为HCC预后生物标志物和治疗靶点的临床应用。
总之,团队的研究,提供了对肝脏TE在调节肝癌基因表达中的潜在作用的见解。此外,研究结果强调了KDM1A通过肝脏TE介导的机制,在肝癌细胞生长中的关键功能,特别是通过抑制HNF4A的转录活性。总体而言,本研究增强了对肝脏TE反式激活活性精确调控的理解,从而为确定肝癌的新治疗靶点开辟了途径。
 腾讯登录
腾讯登录









还没有人评论,赶快抢个沙发