【Science子刊】中山医院赵兴明团队:噬菌体-细菌基因交换,驱动人类肠道结构变异
| 导读 | 在本研究中,团队探索了肠道phageome中潜在的精细遗传变异,尤其是结构变异(SVs)。 |
2024年8月14日,复旦大学附属中山医院、复旦大学类脑智能科学与技术研究院赵兴明团队在期刊《Science Advances》上发表了题为“Long-read sequencing reveals extensive gut phageome structural variations driven by genetic exchange with bacterial hosts”的研究论文。在本研究中,团队使用单分子实时(SMRT)长读长测序,在富含VLPs的宏基因组样本中系统地对SV进行基因分型,揭示了噬菌体SVs在人类肠道病毒组中的流行率。

https://www.science.org/doi/10.1126/sciadv.adn3316
研究介绍
01
噬菌体(包括噬菌体和古细菌病毒)在人类肠道中普遍存在,在调节细菌群落(细菌组)和促进细菌之间的水平基因转移(HGT)方面,发挥着关键作用 。人肠道噬菌体群落(phageome)主要由尾部双链DNA噬菌体(dsDNA噬菌体)组成,包括普遍存在的crAssphage和Gubaphage。噬菌体微多样性在噬菌体生态学中,起着至关重要的作用;其检测对于理解噬菌体的适应、进化,以及噬菌体-细菌相互作用的动力学,至关重要 。
单核苷酸变异(SNV)和结构变异(SV)都是噬菌体微多样性的主要贡献者。随着时间的推移,大多数噬菌体重叠群中,SNV会逐渐积累。此外,在噬菌体群体中,检测到明显的微异质性,其中每种噬菌体类型在任何特定时间点,都由多个噬菌体菌株的复合物表示。
SV是可变的基因组片段,可能包含表型特征(如毒力、宿主免疫逃逸和致病性)背后的功能基因 。与改变单核苷酸的多态性不同,SV包含更大的基因组改变,并可能导致更实质性的功能变化,这可能为噬菌体提供一种快速适应新宿主和逃避宿主防御的方法。它们提供了噬菌体功能的亚基因组分辨率,并且,可能比SNV发挥更显著的作用。迫切需要根据SV对肠道形态进行分析,以更好地了解噬菌体的进化和多样化。长读长测序技术(如Pacific Biosciences(PacBio)或Oxford Nanopore Technologies)可以促进这种分析,这些技术能够覆盖大基因组区域,因此,在SV检测方面,具有很大的优势 。
在本研究中,团队利用病毒样颗粒(VLP)富集的91个个体的长读长测序,对人类肠道语相中SV进行了全面的表征。团队在9,401个宏基因组组装的高质量肠道DNA噬菌体中,发现了14,438个非冗余噬菌体SV。温带噬菌体表现出与细菌宿主进行基因交换的高度倾向,与有毒噬菌体相比,其导致更高的SV密度。研究结果揭示了噬菌体SVs的功能能力,并有助于深入了解人类肠道形状的遗传变异。
研究进展
02
B-to-P HGT事件参与噬菌体SV的形成
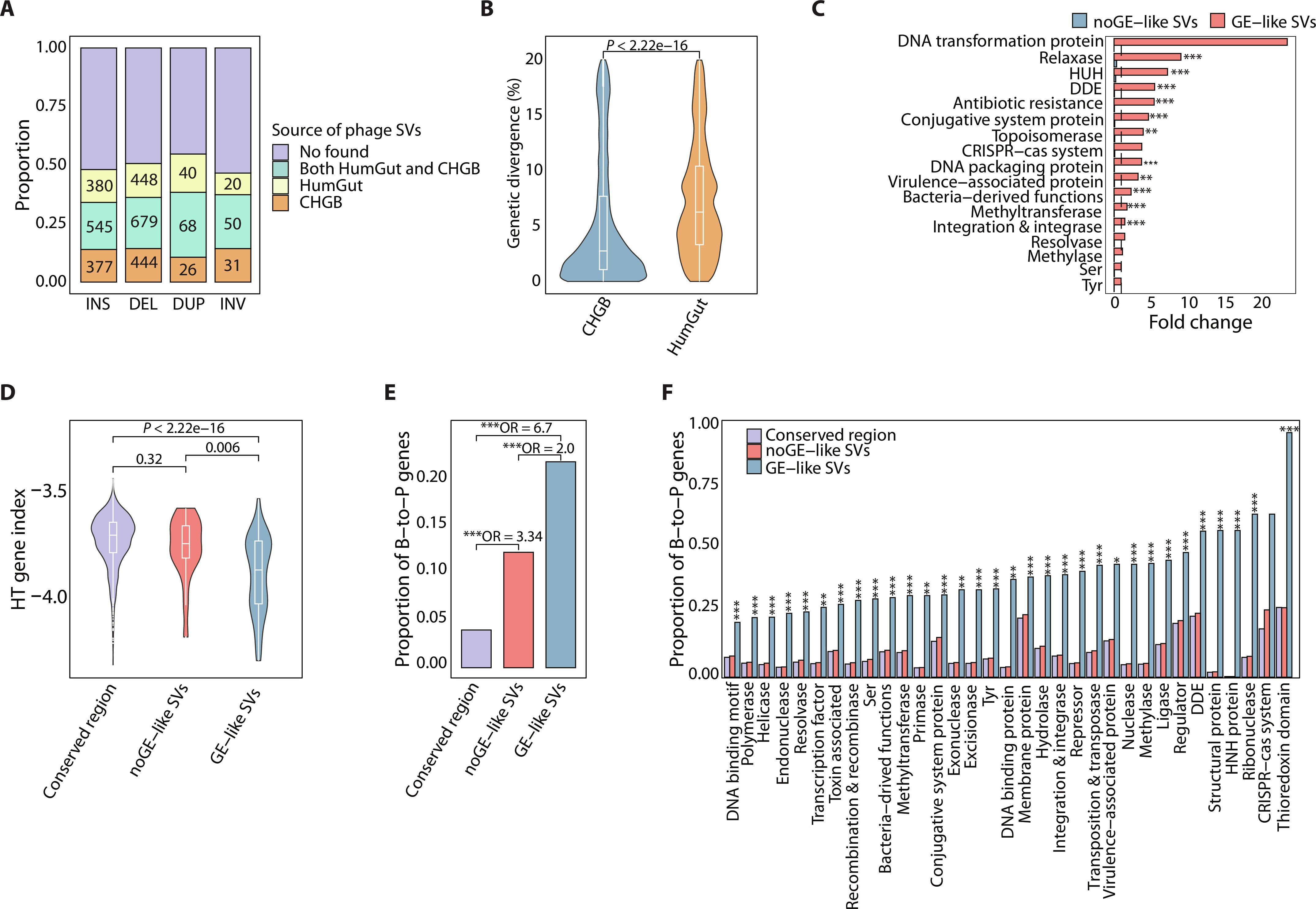
在GE样噬菌体SVs中,大多数(71%;2220个SVs),与从同一队列获得的CHGB基因组匹配。尽管其中 43%(1342 个 SV)表现出与CHGB和HumGut基因组的比对,但是,与HumGut数据集相比,这些SV序列与CHGB基因组的遗传差异,显著降低。这一发现表明,从CHGB 基因组,而不是HumGut基因组,获得了类似GE的SV序列。
与相同基因组的其他区域相比,GE样SVs表现出最高的差异性,支持它们来自不同的基因组。已知的HGT相关基因,特别是DDE和HUH重组酶,以及其他HGT相关基因,包括松弛酶和整合酶,在GE样SVs中显著富集,而不是noGE样SVs。此外,这些GE样SV中有约42% (1341 SV) 携带细菌衍生的基因(从CheckV注释推断)。
团队共鉴定了33,572个B-to-P和79,033个噬菌体-细菌(P-to-B)HGT事件。B-to-P事件,而不是P-to-B事件,在含有GE样SV的区域显著富集,支持这些GE样噬菌体SVs的细菌来源。位于GE样SV区域内的大多数重组酶和转座酶,来源于B-to-P HGT事件,其中约60%的GE样SVs携带的DDEs,被鉴定为B-to-P HGT基因。
这些基因转移事件,有可能促进微生物性状在各个王国之间的传播。团队还发现了涉及CRISPR相关cas1蛋白的B-to-P基因转移事件的几个实例。这些发现证实了,B-to-P基因交换,参与噬菌体SVs的形成。
噬菌体基因交换是噬菌体SVs形成的驱动力。
特定噬菌体-细菌对之间GE样SV序列的基因交换
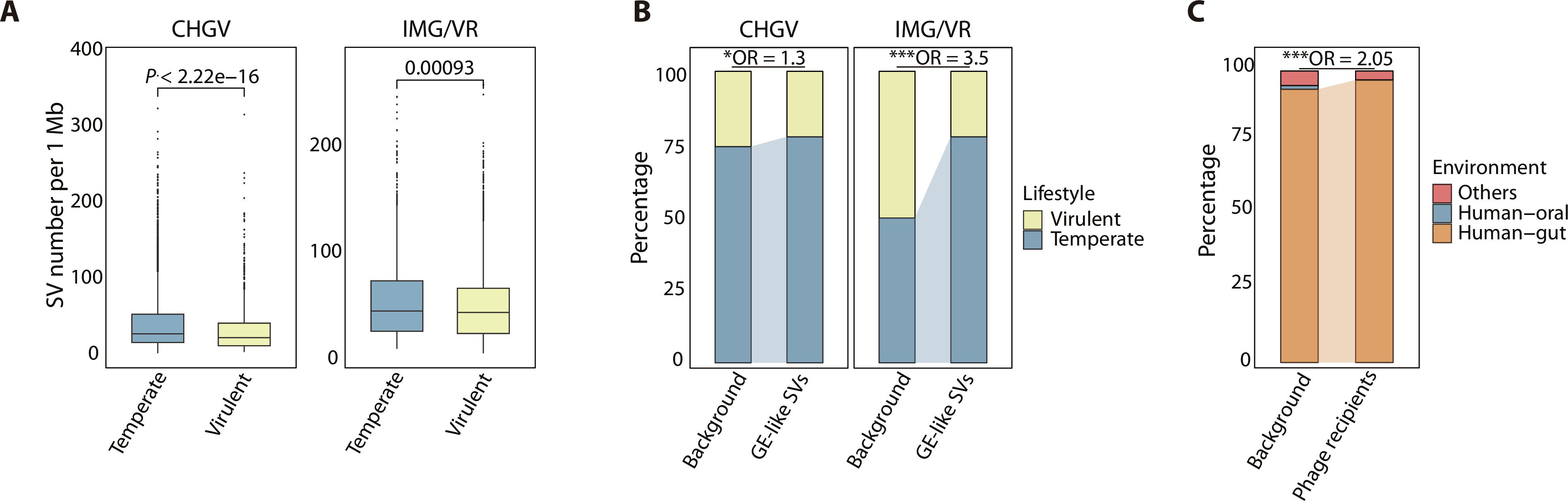
在推断的噬菌体-宿主对中,观察到的GE样SV序列同源性,发生率显著更高(约73%),表明它们之间可能进行序列传输。同一噬菌体重叠群内的这些GE样SV序列,主要与特定的细菌属(37%)或物种(35%)有关。更宽宿主范围的噬菌体重叠群,表现出更高的SV密度。共享GE样SVs的噬菌体-细菌对,表现出显著的正相关关系,并且,这些噬菌体-细菌相关性的强度,随着它们之间共享SV序列的数量而增加。
多种噬菌体物种,从一组有限的细菌中,获得基因组片段以形成SV;而少数噬菌体可以从广泛的宿主中,接收 DNA 序列。细菌和噬菌体之间存在共享的GE样SV序列,并不一定意味着,它们之间存在直接的基因转移。相反,更合理的是,这些共享序列是由中间基因组或其他移动遗传元件促进的,统称为“共享移动元件库”。
温带噬菌体的SV密度,明显高于有毒噬菌体。两种噬菌体生活方式,在含噬菌体SV的区域中,都显示出更高比例的细菌衍生基因。而温带噬菌体SVs内,细菌基因的患病率,显著高于致病性噬菌体中的细菌基因患病率(温带噬菌体:34.1%;致力噬菌体:18.3%)。GE样SVs在温带噬菌体中富集,表明温带生活方式的噬菌体,比具有毒性的噬菌体,更频繁地与细菌宿主进行基因交换。核酸内切酶通过降解宿主细菌的基因组DNA ,在裂解生命周期中发挥关键作用,这与致毒噬菌体的特征性裂解生命周期一致。
GE样SV序列在特定的噬菌体-宿主对之间转移,在温带噬菌体中观察到显著的患病率。鉴定出的GE样SV序列,主要在位于同一生态位的噬菌体和细菌之间共享,特别是人类肠道,这表明生态学是塑造SVs的重要因素。总之,研究结果强调了噬菌体-宿主相互作用,在噬菌体多样化中的关键作用,及其与噬菌体进化的相关性。
不同生活方式对噬菌体SVs的影响。
研究结论
03
噬菌体-细菌基因交换,已被确定为噬菌体SV形成的主要贡献者。然而,噬菌体之间发生的基因转移事件,也可能有助于噬菌体SV的形成。具体而言,噬菌体组装过程中DNA包装过程中的错误,可能导致来自其他近谱系噬菌体基因组的DNA片段,掺入新的病毒粒子中。在同时感染同一细菌细胞的远亲噬菌体之间,观察到基因转移 。噬菌体SVs也可能源于噬菌体和质粒之间的双向遗传交换,而不是细菌染色体。先前的一项研究表明,噬菌体-质粒同时具有质粒和噬菌体的特征,可以介导噬菌体和质粒之间的基因交换。此外,定植于人类肠道的非细菌微生物(即古细菌、真核生物和原生生物),有可能成为肠道病毒的宿主。病毒与这些微生物之间的基因交换,也可以促进病毒SVs的形成。因此,需要对噬菌体内部或噬菌体与其他微生物之间的基因转移,进行更详细的分析,以更好地了解噬菌体多样化的原因和后果。
噬菌体生活方式与SVs的功能分布存在关联,表明进化轨迹可能存在分歧。与有毒噬菌体相比,甲基转移酶在与温带噬菌体相关的SVs中,表现出更高的富集度,这是温带噬菌体的特征。之前的一项研究,阐明了甲基转移酶在温带噬菌体生命周期中的关键作用,它们在帮助噬菌体逃避宿主细菌的限制性修饰系统方面,发挥着关键作用。这种逃避策略使温带噬菌体能够在溶原生命周期中持续存在,从而与其宿主基因组,保持长时间的关联。因此,这提高了噬菌体和细菌宿主之间水平遗传交换的可能性,与具有毒性生活方式的噬菌体相比,导致SV密度升高。温带噬菌体SVs中,切除酶和分离酶显著富集。相比之下,对于有毒噬菌体,核酸内切酶在SVs中显著富集。因此,在温带噬菌体和有毒噬菌体之间观察到的SV功能变化,强调了它们不同生命周期背后的复杂适应。
团队通过关键字搜索,手动将噬菌体蛋白组织到各种功能类别中。请注意,这种将各种生物学功能,聚合为广泛类别的功能注释,可能会影响某些功能的某些蛋白质的检测。例如,尾蛋白在噬菌体的感染过程中发挥重要作用,并表现出快速进化特性,使它们能够适应宿主环境的变化,并逃避宿主免疫反应。尾蛋白发挥不同的功能作用,包括尾部补全、尾部终止子、尾部纤维、尾管等。团队目前的功能注释,无法区分这些不同类型的尾部蛋白。未来,团队将改进功能注释,以便能够检测某些功能的噬菌体蛋白。
参考资料:
1.S. Benler, N. Yutin, D. Antipov, M. Rayko, S. Shmakov, A. B. Gussow, P. Pevzner, E. V. Koonin, Thousands of previously unknown phages discovered in whole-community human gut metagenomes. Microbiome 9, 78 (2021).
2.M. Touchon, J. A. Moura de Sousa, E. P. Rocha, Embracing the enemy: The diversification of microbial gene repertoires by phage-mediated horizontal gene transfer. Curr. Opin. Microbiol. 38, 66–73 (2017).
 腾讯登录
腾讯登录









还没有人评论,赶快抢个沙发