【Lancet子刊】中山大学赵慧英团队:无监督学习方法或揭示脑部疾病高风险基因
| 导读 | 本研究提出了一个贝叶斯框架,通过整合长程染色质相互作用,来预测受先导SNP影响的基因,包括来自不同发育阶段多种脑细胞类型的Hi-C数据的增强子-启动子相互作用 (EPI),以及使用来自英国生物银行的约500,000个样本确定的SNP-SNP相互作用。 |
2024年8月20日,中山大学孙逸仙纪念医院赵慧英团队在期刊《eBioMedicine》上发表了题为“Accurate identification of genes associated with brain disorders by integrating heterogeneous genomic data into a Bayesian framework”的研究论文。在本研究中,团队将贝叶斯框架应用于6种脑部疾病,预测了一组HRG。HRGs在产前脑发育阶段的表达水平高于产后阶段,可能反映了在脑发育的相对早期阶段,发生了初始神经元损伤。基因共表达模块分析和罕见变异分析表明,与神经退行性疾病相关的HRGs与精神疾病,具有共同的遗传结构。本研究提出的方法,促进了科学界对脑部疾病的理解,并促进了新治疗方法的识别。

https://www.thelancet.com/journals/ebiom/article/PIIS2352-3964(24)00322-0/fulltext#%20
研究背景
01
随着全基因组关联研究(GWAS)的快速发展,越来越多的SNPs被发现与疾病相关。这些GWAS的发现,提供了该疾病的潜在致病SNP,但这些SNPs如何影响基因表达途径或参与疾病产生过程,仍然是一个具有挑战性的问题。
研究人员致力于通过将GWAS位点与表观基因组学和转录组学数据,以及其他基因组学数据相结合,来检测风险基因。团队之前提出了一种风险基因预测因子,rGAT-omics,它整合了基因网络、基因与SNP的距离、远端调控元件和基因表达信息,以识别那些受SCZ相关SNP影响的候选基因。然而,目前所有的方法,都忽略了包括重要的基因组或转录组信息的额外来源。
在本研究中,团队建立了一种无监督学习方法,使用长程chromatin interactions(iGOAT)对风险SNP进行评级,以预测与给定疾病相关的高风险基因。该方法采用贝叶斯框架来整合基因组信息和网络,目的是识别一组最可能的疾病相关基因。团队将该方法应用于精神疾病 [精神分裂症(SCZ)、双相情感障碍(BP)、重度抑郁症(MDD)] 和神经退行性/神经系统疾病 [阿尔茨海默病 (AD)、帕金森病(PD)和偏头痛(MG)],以识别 HRG。iGOAT揭示了许多与脑部疾病相关的基因,这些基因在脑细胞中特异性表达,并富含药物靶点。实验结果证实了MLH1(排名靠前的基因之一)与SCZ的关联,SCZ是一种与神经干细胞(NSC)增殖和分化失调相关的神经精神疾病。这种预测方法在揭示导致特定脑部疾病的潜在生物学途径、发育窗口和细胞类型方面,显示出巨大的潜力。
研究进展
02
与脑部疾病相关的多效性HRG,在大脑发育过程中表现出不同的表达谱
团队分析了多效性HRGs(PRGs),其定义为与两种或多种精神疾病或两种神经系统疾病有关。这些基因集分别被称为psyPRG(91个基因)和dePRG(71个基因)。这些基因在大脑发育过程中的表达谱是不同的。产前阶段(1-7)的psyPRG表达远高于产后阶段(8-15)的表达,在第3-9阶段下降,在第10-12阶段略有增加。相比之下,dePRGs在产后阶段的表达高于产前阶段,在第1-9阶段呈上升趋势,然后在第10-15阶段呈下降趋势。这些结果证明,在胎儿脑发育阶段,与精神疾病相关的HRGs活性升高;而与神经系统疾病相关的HRGs,仅在婴儿阶段后,表现出活性升高。psyPRGs和dePRGs的表达谱,分别在神经元和小胶质细胞中,表达更高 。具体来说,在神经元亚型中,psyPRGs在几乎所有兴奋性神经元中都表现出高表达,而dePRGs则没有;dePRGs在某些抑制性神经元中,表现出高表达。
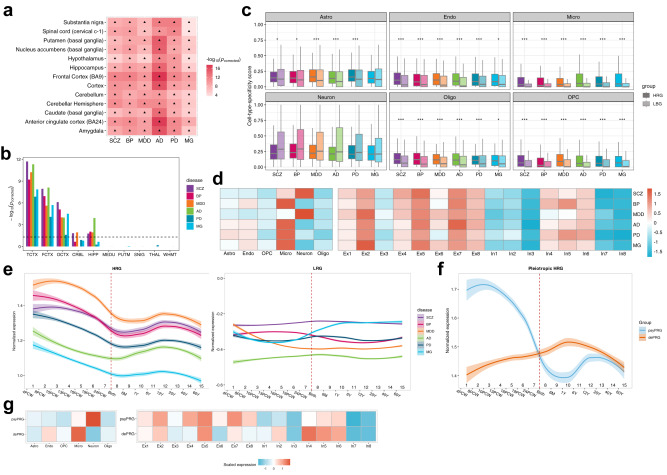
HRGs、LRGs和多效性HRGs的表达分析。(a)使用GTEx数据集,对HRG与LRG的组织特异性分析(方法)。“▴”表示显著结果(Fisher 精确检验 p纠正< 0.05)。(b)使用 BrainEAC的表达谱,对HRG与LRG进行组织特异性分析(方法)。(c)HRGs与LRGs的细胞特异性分析(方法)。右图显示了神经元亚型中HRGs和LRGs的细胞类型表达水平(“∗”表示HRGs的表达)。颜色表示该细胞类型中基因的列尺度平均表达水平。(d)不同细胞类型中HRGs的中位表达水平的比较。颜色表示该疾病中基因的行尺度中位数表达水平。(e)HRGs和LRGs在整个大脑发育阶段的发育表达轨迹。虚线代表出生。阴影的aera,表示表达水平的95%置信区间。(f)psyPRGs和dePRGs在大脑发育阶段的表达轨迹。(g)脑细胞和神经元亚型中,psyPRGs和dePRGs的行尺度平均细胞表达谱。
预测的HRG可能揭示了脑部疾病之间的共同机制
与精神疾病相关的HRG,在与神经退行性疾病相关的失调基因表达模块(MEcyan、MEred 和 MEpink)中富集,反之亦然(MEblack、MEpink、MElightgreen、MEred和MEcyan)。与所有6种脑部疾病相关的HRG,都富含AD患者和对照组之间差异表达(DEGs)的基因。SCZ的DEGs中,富集了与SCZ相关的HRGs。基因表达差异分析鉴定出6个DEGs(|log2(倍数变化)| > 0和t检验pcorrected<0.05]为BP,其中没有BP的HRGs和LRGs。与此类似,共发现79个PD基因,其中3个基因为PD的HRGs。HRGs和DEGs之间用于PD的重叠基因数量(Fisher's exact testp=0.049) 比LRG略高。因此,在神经退行性疾病中失调的模块中,与精神疾病相关的HRGs的富集,可能很好地反映了这些疾病之间共享的神经机制。
团队通过寻找HRGs的富集,进一步探索了6种脑部疾病之间的共同机制,这些HRGs具有与SCZ和ASD相关的罕见变异,以及与发育障碍相关的新发突变(DD)。SCZ、MDD、AD和PD相关的HRG,在已知具有与ASD和DD相关的罕见变异的基因中富集。与AD相关的HRGs,也富集在携带与SCZ相关的罕见变异的基因中。
与本文研究的5种疾病(SCZ、MDD、AD、PD 和 MG)相关的HRG,都在已知携带与ASD相关的HGMD致病变异的基因中富集。鉴定为与SCZ、BP、MDD和AD相关的HRGs,也富集在与SCZ相关的HGMD致病变异中。因此,HRGs所携带的致病变异,可能表明脑部疾病之间,存在共同的遗传结构。
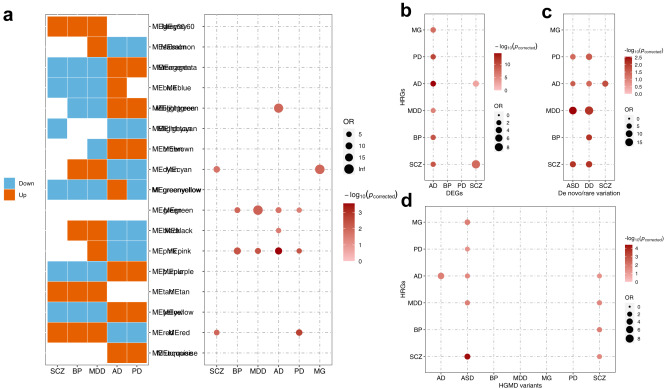
HRGs与6种脑部疾病相关的共同遗传机制。(a)与疾病相关的失调基因模块,在左侧块中显示颜色。与LRG相比,HRG在基因模块中的富集如右侧块所示。“ME”是指模块特征基因。(b)使用LRGs作为背景基因的AD、BP、PD或SCZ患者和对照中差异表达基因(DEG)中HRGs的富集分析。(c)使用LRGs作为背景基因,对携带与ASD、DD或SCZ相关的新发/罕见变异的基因中的HRGs进行富集分析。(d) 以LRGs为背景基因,对携带AD、ASD、BP、MDD、MG、PD或SCZ致病突变的HGMD基因中的HRGs进行富集分析。阴影的强度,表示显著性的程度。圆点的大小,表示单侧Fisher's Exact检验中比值比(OR)的大小,用于评估与LRG相比,基因集中HRG的富集。
研究结论
03
在iGOAT中使用EPI,可以提高其识别脑组织和区域中特异性表达的基因的能力。对于与神经元不无关系的疾病,团队建议用户在使用Github中的数据和代码运行iGOAT时,应排除神经元的EPI。
当iGOAT被应用于精神疾病和神经系统疾病时,它可能揭示了脑部疾病的神经发育起源。AD相关的基因共表达模块(MEpink和MEcyan)都被与BP、MDD或SCZ相关的HRGs富集。与SCZ、BP、MDD、AD、PD和MG相关的HRGs,共计133、133、106、146、146和86个HRGs与AD的差异表达基因(DEG)重叠。与SCZ、BP、MDD和PD相关的HRG,富含与发育障碍(DD)相关的罕见变体。这一发现,可能反映了AD/PD和精神疾病之间共享的遗传结构。
自闭症谱系障碍在遗传上具有高度异质性,可能由遗传变异和新发变异引起。用于MDD的HRG,富含与ASD相关的新生突变。此外,自闭症谱系障碍与MDD有很强的遗传相关性,用于MDD的HRG,富含与DD相关的新生突变。团队还发现,与帕金森病相关的HRG,富集了与ASD相关的新生突变。
总之,本研究提出了一种方法,即iGOAT,它将异质基因组数据集成到贝叶斯框架中,以允许预测疾病相关基因。iGOAT在精神疾病和神经系统疾病中的应用,揭示了EPI和SNP-SNP相互作用在预测中的重要性。iGOAT可以潜在地促进从GWAS研究结果中,得出神经学相关假设。例如,iGOAT可以帮助科学界产生一个假设,即许多与疾病相关的SNPs通过识别HRGs,而不是最接近索引SNPs的基因,远程调节元件发挥作用。此外,iGOAT在广泛使用的已知基因集中,发现了许多未报告与脑部疾病相关的HRG。这些HGR可以帮助科学界产生一个假设,即这些疾病与新的基因网络有关。iGOAT还可以应用于非脑部疾病,方法是将与非脑部疾病相关的SNPs作为输入,并利用来自与特定疾病相关的组织的多组学数据,来构建贝叶斯框架。
参考资料:
1.Buniello A. MacArthur J.A.L. Cerezo M. et al.
The NHGRI-EBI GWAS Catalog of published genome-wide association studies, targeted arrays and summary statistics 2019.
Nucleic Acids Res. 2019; 47: D1005-D1012
2.Zhao H. Yang Y. Lu Y. et al.
Quantitative mapping of genetic similarity in human heritable diseases by shared mutations.
Hum Mutat. 2018; 39: 292-301
 腾讯登录
腾讯登录









还没有人评论,赶快抢个沙发