【Nature子刊】中国医科大学宫月华团队:细胞衰老相关基因与多种癌症风险的潜在因果关系
| 导读 | 在这项研究中,团队确定了5个CSR基因(CNOT6、DNMT3B、MAP2K1、TBPL1和SREBF1)、18个DNA甲基化基因和LAYN蛋白表达,它们都与不同的癌症类型有因果关系。 |
2024年8月31日,中国医科大学第一临床学院宫月华团队在期刊《Communications Biology》上发表了题为“Mendelian randomization reveals potential causal relationships between cellular senescence-related genes and multiple cancer risks”的研究论文。研究结果表明,以遗传易感性为特征的CSR基因,在各种肿瘤中发挥因果效应。团队确定了5个基因表达水平、18个基因DNA甲基化模式和1个蛋白质表达特征,这些特征与癌症风险有因果关系,并且已经针对4种蛋白质,开发了靶向药物。其余基因或甲基化位点,代表了未来针对癌症的小分子药物开发研究的潜在靶标。这项研究将遗传位点、基因表达、DNA甲基化和蛋白质表达与不同的癌症联系起来,即遗传变异可能通过影响基因表达、甲基化水平或蛋白质表达,来影响肿瘤发生。这为探索潜在的肿瘤发生机制和逆转疗法,提供了有力的证据和有前途的应用。

https://www.nature.com/articles/s42003-024-06755-9
研究介绍
01
细胞衰老是对不同外部刺激的生物反应,包括微环境压力、营养剥夺、DNA损伤、细胞和细胞器损伤,以及细胞信号网络异常。该过程表现为四个特征:细胞周期停滞、衰老相关分泌表型(SASP)、大分子损伤和代谢紊乱。细胞衰老是最近假设的分子癌症标志,对与年龄相关的疾病、致癌作用和组织修复,具有重要意义。
目前,基于传统的观察性研究,已经确定了特定细胞衰老相关(CSR)基因与多种肿瘤风险之间的关联。例如,EZH2和TOP2A敲低通过诱导细胞衰老,来抑制肝癌发生。IL-1是SASP的关键成分,可促进口腔前体病变的恶性转化。SOX6通过诱导细胞衰老,来抑制宫颈细胞增殖。此外,FBXW7可能通过调节细胞衰老和DNA损伤修复等机制,在食管鳞状癌中起肿瘤抑制因子的作用。
孟德尔随机化(MR)位于观察性研究和RCT之间的交叉点,在支持观察性试验中的因果证据方面越来越重要。MR使用遗传变异作为IVs,来研究终生暴露与结果之间的潜在因果关系。MR已被广泛用于审查各种性状和疾病之间的因果关系,例如,脂肪酸和精神分裂症,以及肺功能和心血管疾病。基于汇总数据的MR(SMR)和双样本MR(TSMR)都能够估计遗传决定的性状(例如,基因表达、DNA甲基化和蛋白质表达)与感兴趣的复杂性状(作为结果的疾病表型,例如,肿瘤)之间的多效性或潜在因果关系。
在这项研究中,团队使用SMR或TSMR,分析了18种癌症中CSR基因表达、DNA甲基化和蛋白质表达水平之间的潜在因果关系。通过贝叶斯共定位,团队深入了解了支撑癌症风险变异的潜在调节机制。团队还探讨了已鉴定的CSR基因的成药性和功能作用。
研究进展
02
具有多种癌症风险的细胞衰老全基因组顺式eQTL的MR和共定位分析
团队确定了乳腺癌中4个独特遗传位点的4个关联信号,结直肠癌中2个独特基因位点的2个关联信号,子宫内膜癌中1个独特遗传位点的1个关联信号,肺癌中2个独特基因位点的2个关联信号,黑色素瘤中2个独特遗传位点的2个关联信号, 以及前列腺癌中16个独特遗传位点的16个关联信号。除上述情况外,团队未发现CSR基因表达与其他12种癌症类型之间,存在任何显著的遗传联系。
5个基因的表达,表现出与某些癌症相同的因果变异。这些基因分别是患有肺癌的CNOT6,以及患有前列腺癌的DNMT3B、MAP2K1、TBPL1和SREBF1。对于肺癌,CNOT6表达的SD增加1次,与肿瘤风险增加18%相关(比值比OR:1.18,95%置信区间CI):1.10–1.27,FDR=3.46×10−2)。对于前列腺癌,DNMT3B表达的SD增加1次,与风险增加73%相关(OR:1.73,95%CI:1.48–1.97,FDR=1.49×10−3),MAP2K1表达的SD降低1次,与风险降低24%相关(OR:0.76,95%CI:0.66–0.86,FDR=5.82×10−6),TBPL1表达的SD降低1次,与降低16%的风险相关(OR:0.84,95%CI:0.76–0.92,FDR=2.81×10−3),SREBF1表达的SD降低1次,与风险降低11%相关(OR:0.89,95%CI:0.85–0.94,FDR=4.82×10−4).
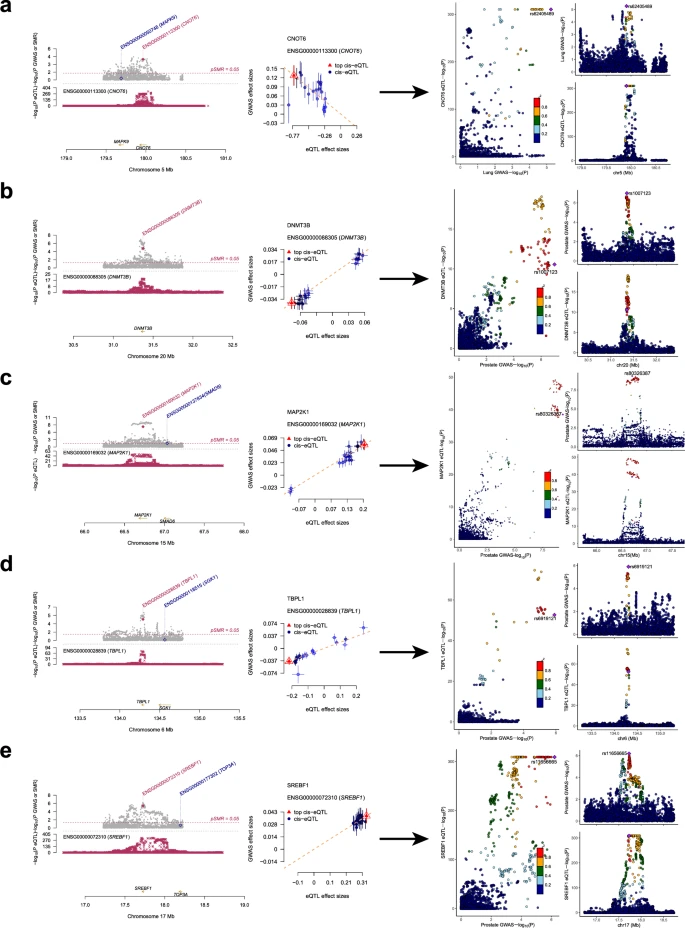
SMR和共定位分析,优先考虑CSR基因和癌症风险。
具有多种癌症风险的细胞衰老全基因组顺式-mQTL的MR和共定位分析
团队确定了结直肠癌中3个独特基因位点的3个关联信号,子宫内膜癌中5个独特基因位点的5个关联信号,肺癌中10个独特基因位点的10个关联信号, 口腔咽部3个独特遗传位点的5个关联信号,前列腺癌中115个独特遗传位点的152个关联信号,胃癌中3个独特基因位点的4个关联信号,甲状腺癌中的1个关联信号。团队没有发现CSR基因、DNA甲基化和其他癌症类型之间,有任何显著的遗传关联。
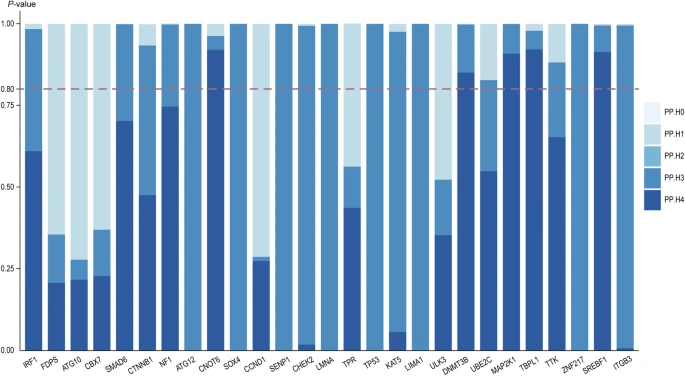
贝叶斯共定位分析确定了18个基因,这些基因在DNA甲基化和癌症之间,表现出共同的因果差异。此外,这些分析表明,调节这些基因的不同遗传变异,对甲基化水平产生不同的影响,从而影响观察到的结果。
在结直肠癌中,NTN4和SMAD6高甲基化与风险增加相关(OR:1.25,95%CI:1.15–1.35,FDR=1.77×10−2和OR:1.31,95%CI:1.18-1.44,FDR=4.52×10−2)。在子宫内膜癌中,NF1高甲基化与风险增加相关 (OR:1.34,95%CI:1.21–1.48,FDR=2.62×10−2),而TERT高甲基化与风险降低相关(OR:0.78,95%CI:0.66–0.90,FDR=4.16×10−2)。对于肺癌,ATG12高甲基化(OR:0.92,95%CI:0.88–0.96,FDR=7.43×10−3)、DDAH2高甲基化(OR范围为0.65至0.75,FDR<0.05)、ENO1高甲基化(OR:0.89,95%CI:0.83–0.95,FDR=1.62×10−2)和TP53BP1高甲基化(OR:0.80,95%CI:0.70–0.90,FDR=7.07×10−3) 都降低了肿瘤风险。对于口腔咽部,KCNJ12高甲基化与风险增加相关(OR范围为1.17至1.23,FDR=4.38×10−2),而NOTCH1高甲基化降低了肿瘤风险(OR:0.51,95%CI:0.21–0.82,FDR=4.38×10−2)。在前列腺癌中,同一基因内的不同甲基化位点,可能产生不同的影响。与DNMT3B中rs2424905、rs6087989、rs1883730、rs1003521和rs4911106相关的6个CpG位点中的甲基化位点cg26553763和cg13636640,与肿瘤风险呈负相关 (OR:0.83,95%CI:0.75–0.92,FDR=3.47×10−3和OR:0.96,95%CI:0.94–0.97,FDR=1.41×10−4),而cg22052056、cg21235334、cg13788819和cg09149842与肿瘤风险呈正相关(OR:1.15,95%CI:1.09–1.22,FDR=1.07×10−3;OR:1.13,95%CI:1.07–1.18,FDR=1.03×10−3;OR:1.12,95%CI:1.07–1.17,FDR=5.72×10−4;和OR:1.14,95%CI:1.08–1.19,FDR=7.78×10−4)。同样,FERMT1和MAD1L1中不同甲基化位点,对前列腺癌风险的影响为阳性或阴性;MVK、NEK6、NUDT5和SMAD2高甲基化降低了肿瘤风险(OR:0.91,95%CI:0.86–0.95,FDR=3.47×10−3;OR:0.96,95%CI:0.95–0.98,FDR=6.28×10−4;OR:0.96,95%CI:0.94–0.98,FDR=1.40×10−3;和OR:0.93,95%CI:0.90–0.96,FDR=3.36×10−3)和SPI1高甲基化增加了肿瘤风险(OR:1.15,95%CI:1.08–1.21,FDR=3.30×10−3)。对于甲状腺癌,SMAD3高甲基化与肿瘤风险增加相关 (OR:1.69,95%CI:1.47–1.92,FDR=1.73×10−2)。
此外,特定CSR基因中DNA甲基化模式的改变,对其mRNA表达有直接影响。研究结果表明,TERT、DDAH2、DNMT3B、MAD1L1、NOTCH1、 TP53BP1、 MAP2K1、SMAD2、 SMAD3、SMAD6和SREBF1的DNA甲基化,显著影响其基因表达水平。
具有多种癌症风险的细胞衰老全基因组cis-pQTL的MR和共定位分析
LAYN蛋白表达水平升高与前列腺癌风险降低有关(OR:0.51,95%CI:0.37-0.71,FDR=1.36×10−2),并与这种疾病共享一个因果变异。
eQTLs和GWAS的共定位结果。
研究结论
03
总之,团队使用MR分析,确定了CSR基因对几种癌症的潜在因果效应,这表明细胞衰老机制在肿瘤发生中起重要作用。进一步探索肿瘤中支撑这些CSR基因的机制,可能有助于预防癌症和加速治疗。此外,这项研究为进一步探索基于CSR基因的抗癌临床药物,提供了坚实的理论基础。
参考资料:
1.Lee, S. & Schmitt, C. A. The dynamic nature of senescence in cancer. Nat. Cell Biol. 21, 94–101 (2019).
2.Gorgoulis, V. et al. Cellular Senescence: Defining a Path Forward. Cell 179, 813–827 (2019).
 腾讯登录
腾讯登录









还没有人评论,赶快抢个沙发