【Nature子刊】中国科学院李鑫团队:创新开发AI跨物种基础模型
| 导读 | 在这项研究中,团队构建了一个包含超过1.2亿个人类和小鼠单细胞转录组的广泛数据集,并开发了一个以知识为依据的跨物种基础模型,名为GeneCompass。 |
2024年10月8日,中国科学院动物研究所李鑫团队在期刊《Cell Research》上发表了题为“GeneCompass: deciphering universal gene regulatory mechanisms with a knowledge-informed cross-species foundation model”的研究论文。在这项研究中,GeneCompass展示了使用人工智能技术破译通用基因调控机制的优势,并显示出加速发现关键细胞命运调节因子和候选药物靶标的巨大潜力。
https://www.nature.com/articles/s41422-024-01034-y
关于GeneCompass
01
随着组学测序技术的快速发展,科学界已经开始剖析各种器官中的细胞如何在单细胞分辨率下发挥其特定功能,从而积累了大量的单元数据。然而,基因表达在多个水平上受到调节,从染色质可及性到转录后修饰。
近年来,一些研究利用单细胞转录组数据构建了预训练的基础模型,例如,scGPT、基因形成者,UCE和scFoundation。这些研究的共同点,是利用数千万个人类单细胞转录组图谱来预训练基础模型,并在广泛的下游任务中表现出卓越的性能,例如,细胞聚类、细胞类型注释、基因扰动模拟和药物靶点预测。
在这项研究中,团队提出了GeneCompass,这是一个在scCompass-126M上预先训练的知识后跨物种基础模型。scCompass-126M是最大的语料库,经过数据预处理,使用了101,768,420个细胞。GeneCompass在不同生物环境中实现了优于最先进的(SOTA)模型的性能,甚至与之相当。该模型可用于剖析从小鼠到人类的通用基因调控机制,并加快确定细胞命运的关键调节因子和药物开发的潜在靶标。
GeneCompass的架构和预训练
02
GeneCompass是一个以知识为基础的跨物种基础模型,在超过1.2亿个人类和小鼠细胞的转录组语料库上,进行了预训练。4种类型的生物学知识(GRN、启动子信息、基因家族注释和基因共表达关系)被整合到GeneCompass的自我监督预训练中。利用自我注意机制进行显式上下文编码,GeneCompass可以根据输入转录组,了解细胞的本质和基因之间的复杂关系。预训练的GeneCompass,旨在通过进一步微调有限的特定任务数据,有效地应用于各种下游生物任务。

GeneCompass架构和预训练语料库。
GeneCompass支持细胞命运预测并识别关键调控因子
03
与低水平过表达OSKM的细胞相比,OSKM高水平过表达的细胞进一步向iPSC状态移动,反映了细胞重编程的精确模拟。所有模拟的敲除细胞,都表现出向内胚层状态的转变。移位程度与计算机敲除水平之间,存在正相关。
团队确定了前5个基因,即NR2F1、NR5A1、WT1、TCF21和GATA4,其模拟细胞包埋与性腺祖细胞和成熟Leydig细胞的相似性较高,而与原始ESC的相似性较低。这5个基因都是转录因子,其中3个,即WT1、NR5A1和NR2F1,据报道对小鼠体内性腺发育至关重要。
NR5A1和GATA4过表达细胞,直接上调了参与维持视黄酸稳态的RBP1基因和参与睾丸细胞中类固醇合成的相关基因STAR和HSD3β1。这些结果表明,预测的基因确实在与人类ESC的性腺分化中发挥了重要作用。
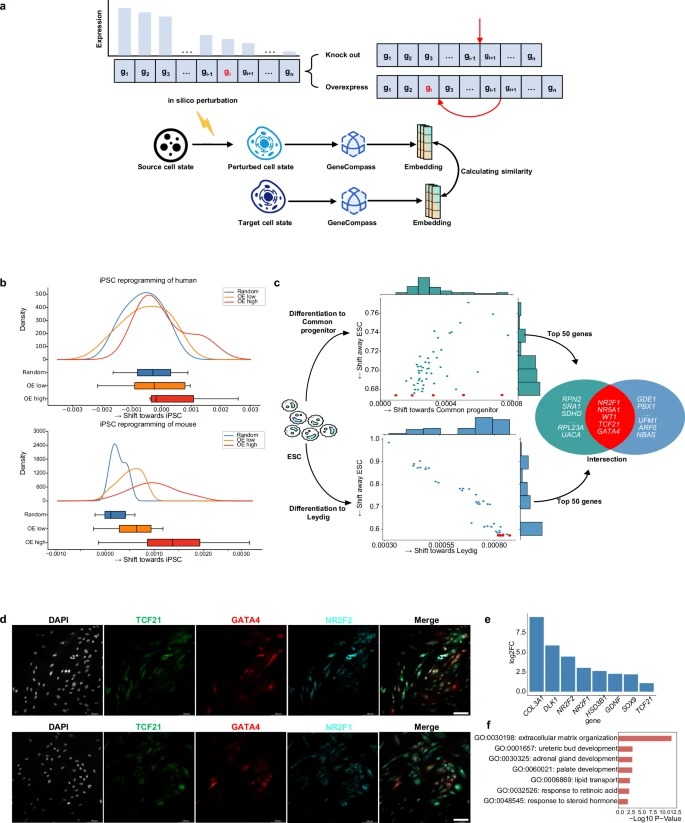
用于细胞重编程和分化的计算机定量扰动。
结论
04
GeneCompass仍有改进的潜力。该模型仅包含来自两个物种的信息:人类和小鼠。当尝试纳入来自其他物种的数据时,团队怀疑,物种特异性基因表达模式,可能会抵消扩大数据量的好处。除了当前的先验知识外,还应探索其他基本信息,例如,增强子和蛋白质序列。此外,除了单细胞水平的转录数据外,大量的表观基因组学、蛋白质组学和代谢组学数据,将为基因调控提供更丰富的见解。将多模态信息整合到模型中的有效策略,是未来研究的关键途径。
GeneCompass在多项下游任务中表现出良好的性能。随着不断发展和使用率的增加,它有望在优化细胞命运预测和揭示关键调控因素方面提供巨大价值。这可以为其临床应用开辟新的途径,例如,疾病靶点基因发现、肿瘤药物筛选和药物毒性预测。未来,团队预计,大型基础模型和湿实验的融合,将为生命科学研究创造一种新的范式,促进各个领域的进步。
参考资料:
1.Almanzar, N. et al. A single-cell transcriptomic atlas characterizes ageing tissues in the mouse. Nature 583, 590–595 (2020).
2.Regev, A. et al. The human cell atlas. Elife 6, e27041 (2017).
 腾讯登录
腾讯登录









还没有人评论,赶快抢个沙发