【Nature】清华大学陈柱成/李雪明合作揭示NuA4选择性乙酰化组蛋白H4的机理
| 导读 | 生物体遗传信息DNA缠绕组蛋白八聚体1.7圈形成了核小体——染色体的基本组成单位。组蛋白 H4 的 N 末端与附近的核小体相互作用,促进高级染色质结构的形成和异染色质沉默。酵母中的 NuA4 及其在哺乳动物细胞中的同系物 Tip60 复合物是催化 H4 乙酰化的关键酶,具有调节染色质包装、转录激活和 DNA 修复功能。 |
10月5日,清华大学生命科学学院陈柱成教授与李雪明副教授团队在《Nature》杂志发表题为“Structure of the NuA4 acetyltransferase complex bound to the nucleosome”的研究论文,揭示了乙酰转移酶NuA4结合核小体以及组蛋白H4空间识别的机理,阐明了NuA4作为转录共激活因子发挥功能的结构基础。

https://www.nature.com/articles/s41586-022-05303-x
研究概括
研究人员报告的冷冻电子显微镜结构的NuA4从酿酒酵母绑定到核小体。NuA4 包含两个主要模块:催化组蛋白乙酰转移酶 (HAT) 模块和转录激活因子结合 (TRA) 模块。核小体主要与 HAT 模块结合,并定位在接近多基表面的 TRA 模块,这对于 NuA4 的最佳活性很重要。携带上游激活序列的核小体连接器 DNA是面向保守的,转录激活因子结合表面的Tra1 亚基,这表明 NuA4 作为转录共激活因子的潜在机制。HAT 模块通过 H2A-H2B 酸性斑块和核小体 DNA 识别核小体的圆盘面,将 Esa1 的催化口袋投射到 H4 的 N 端尾部并支持其在 H4 选择性乙酰化中的功能。总之,我们的研究结果说明了NuA4是如何组装的,并为核小体识别和转录共激活的HAT提供了机制见解。
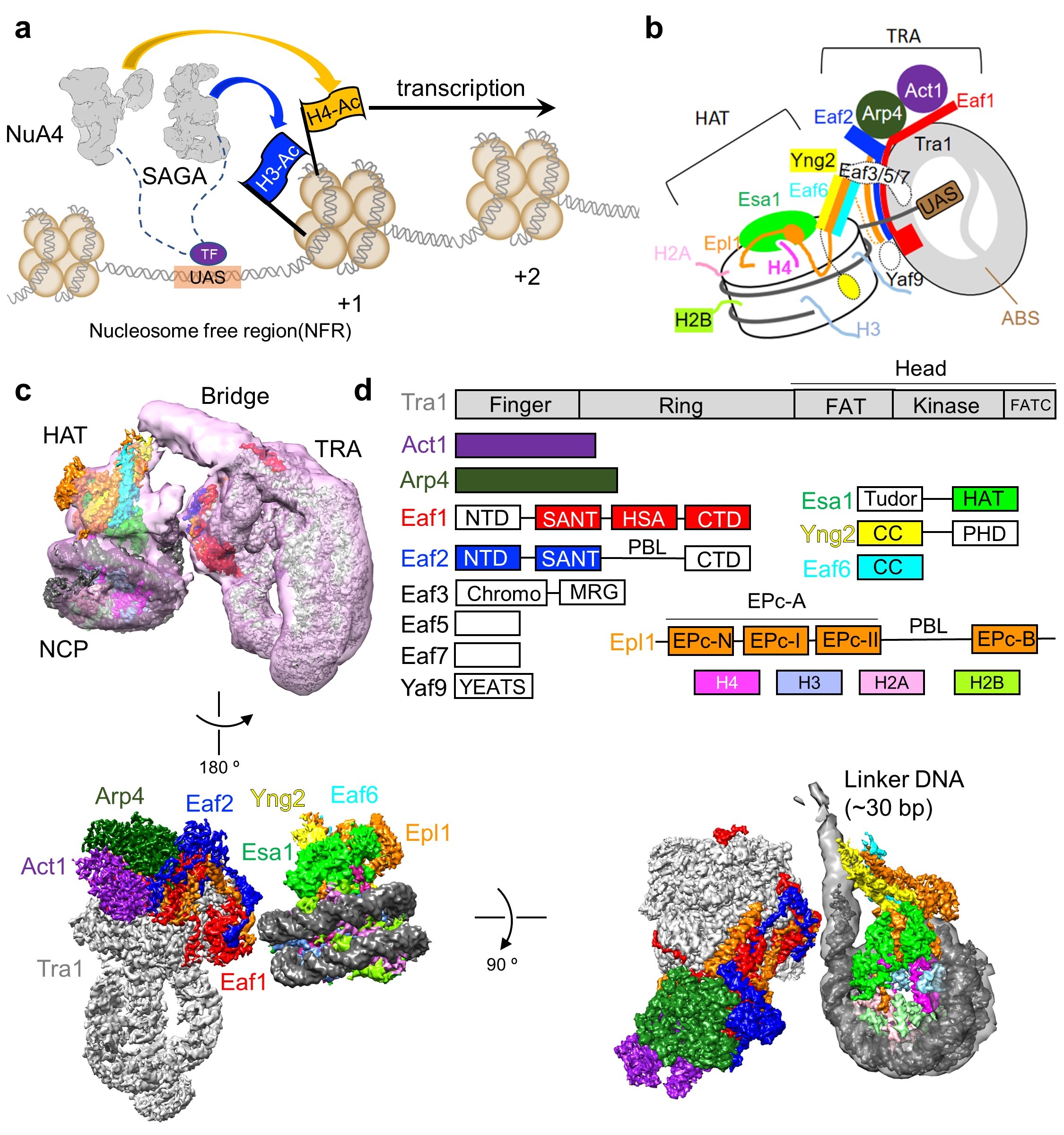
研究进展
结构显示,携带UAS的linker DNA延伸向Tra1亚基表面。研究人员发现该表面在不同物种中高度保守,且众多转录因子与Tra1的结合位点均位于该表面内。因此,该保守面被命名为转录因子结合面(transcription factor binding surface, ABS)。ABS的发现提供了NuA4被转录因子招募至启动子区域的结构基础。
在ABS外周,研究人员发现一个多碱性氨基酸界面(poly-basic surface, PBS)与核小体的linker DNA相互作用。研究人员通过生化以及遗传学实验验证了PBS的对乙酰转移活性,以及调控酵母碳源代谢的重要性。

催化中心HAT模块通过两个关键元件结合在核小体盘状结构表面。其中,Epl1的“arginine anchor”识别核小体的酸性区,“double function loop (DFL)”识别核小体的超螺旋1.5位置处DNA。该核小体识别模式使得Esa1的活性口袋恰好处在H4 的N端尾巴上方。这个结构在全酶水平支持了H4尾巴空间位置识别机制。该机制区别于常见的基于氨基酸序列识别的组蛋白修饰酶工作机制。
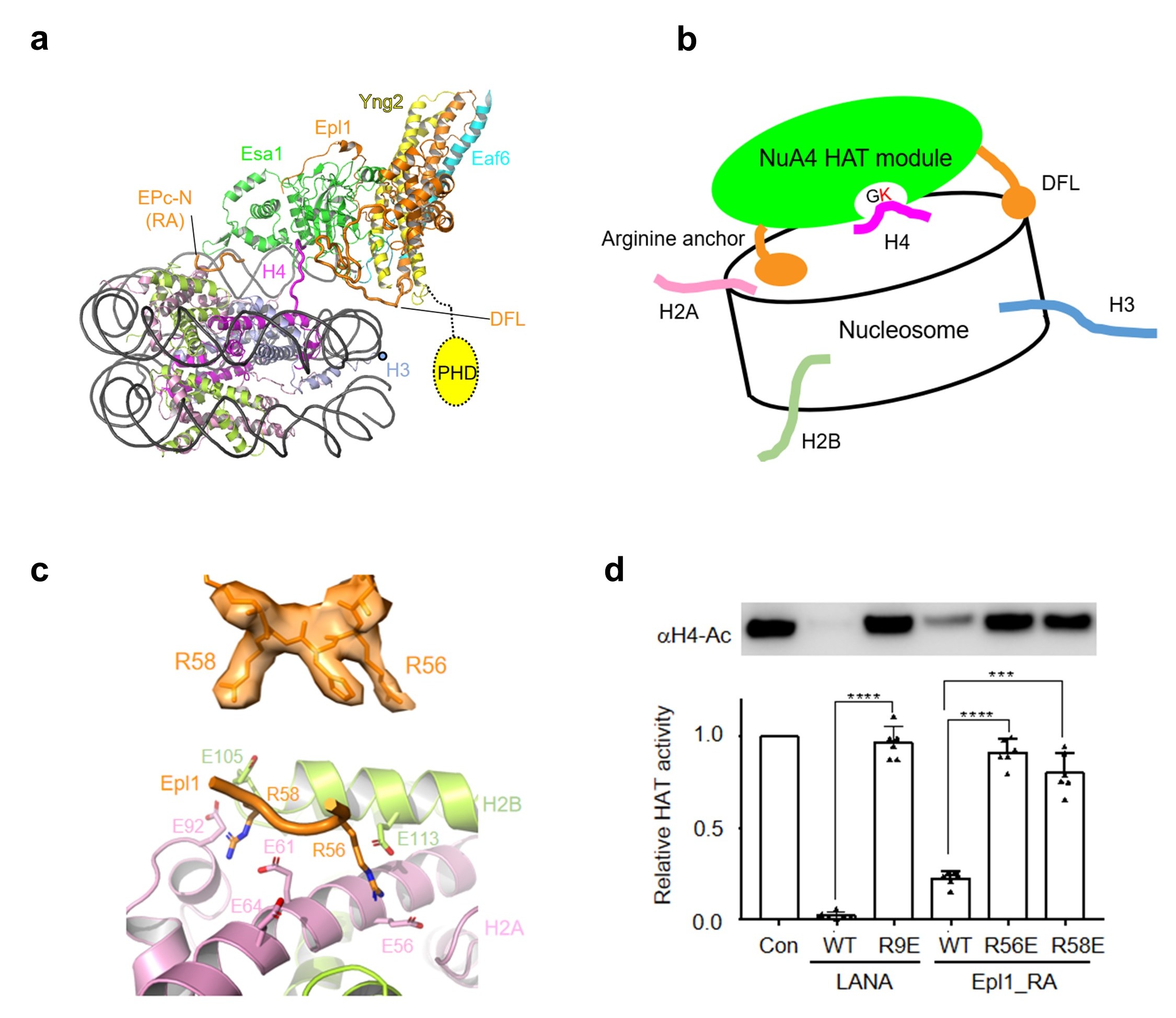
研究意义
该研究揭示了NuA4通过多个结构元件,并协调识别核小体的组织机理。ABS通过转录因子,PBS直接结合接头DNA,招募核小体至TRA模块边缘;此构象使得HAT模块通过DFL和arginine anchor识别核小体的表面,从而发挥选择性乙酰化H4的功能。TRA模块与HAT模块之间的可塑性,使得NuA4能够适应复杂的染色体环境,被不同转录因子招募。除了Eaf5亚基之外,其他亚基在人源TIP60复合物中均存在同源蛋白。因此,该研究对于理解TIP60复合物的组装及工作机制提供了很好的模型。(转化医学网360zhyx.com)
参考资料:
https://www.nature.com/articles/s41586-022-05303-x
https://life.tsinghua.edu.cn/info/1131/4213.htm
注:本文旨在介绍医学研究进展,不能作为治疗方案参考。如需获得健康指导,请至正规医院就诊。
 腾讯登录
腾讯登录


还没有人评论,赶快抢个沙发