智因东方速递| 儿科基因组学:在儿童罕见病诊断中的应用连载(二)
| 导读 | 昨天的推送中论述了传统的基因诊断检测、全外显子测序(WES)或全基因组测序(WGS)在目前临床上应用的主要适应症以及儿科基因组学的发展。今天这篇文章主要介绍儿科基因组学的数据整合和解读。 |
数据的整合和解读
在过去的十年中,越来越清楚的是,个体基因组的信息不能单独被解析。需要了解群体中出现的背景变异以过滤常见变异。理想情况下,这些信息来自随机抽样的个体,并且这些个体没有通过任何特定的临床或社会特征来进行选择,需要成千上万个这样的基因组,对变异进行提取、注释和解读。最佳的变异分类,还需要检测个体详细而全面的临床信息,而不是简单的诊断标签。WES和WGS数据量之大,需要自动化的变异过滤,从而可以集中处理与临床相关的基因组区间和/或变异特征。 用于诊断罕见病的分析工作流程尚未完全标准化,因为实现强大基因诊断的工作流程环节包括原始数据的处理、致病性变异的识别、临床数据的整合等 (FIG. 3),是非常复杂的且仍在不断发展完善中,且因为每一步都有其自身的问题需要进行个体化的评估。
1.变异的提取和注释变异的提取和注释涉及高度复杂的过程,使用同一流程对同一WGS样品进行重复分析,其重复性一般小于100%。 变异提取通过将数据中表示的每个位置与参考基因组中的等同位置进行比较,从而识别出个体等位基因碱基组成的差异。提取的灵敏度取决于检测数据和参考数据的质量及所采用的算法。低质量或低覆盖率的数据会导致输出结果中重要变异的丢失。在重复序列中进行变异提取是非常难的,尤其是对小的插入缺失,这也会导致错误的诊断,尽管变异提取过程发生在原始数据处理阶段。变异提取数据通常保存在标准化的VCF(Variant Call Format,VCF) 文件中,该文件通常被修改包括对每个变异进行注释以用于临床分析。一些注释对于临床解读来说是非常关键的,尤其是次要等位基因频率(Minor Allele Frequencies,MAFs)和变异危害性预测。所有的注释,除了MAF数据, 都会随着注释时所使用的数据和软件工具而变化。因此,尽管具有标准化的结构,来自相同的数据的VCF文件在不同的提供者和不同的版本之间会有很大的差异。
Box 1|儿科基因组学的伦理、法律和社会影响
儿科基因组学同样存在临床遗传学几十年来一直面对的许多伦理、法律和社会问题,例如对生殖自主权、科研项目的知情权、双亲关系的隐私以及对家庭成员的影响。然而,基因组数据量的巨大和广度还带来基因检测本身的新问题,这对儿科检测来说更为复杂;儿童缺少是否接受检测和/或研究的自主权,这意味着在决定什么最符合患儿的利益方面,父母和临床医生起到更重大的作用。基因组学时代的绝大多数新伦理问题都与数据的存储、解读和使用有关。
数据存储 由于临床级别下一代测序(NGS)数据的大小,及对其分析所需的计算能力,应该如何以及在何处存储这些数据尚不清楚。此外,不清楚谁可以利用,或是什么时候应该允许利用这些数据。例如,医疗记录是应该仅包含用于诊断的变异,或是所有潜在的临床相关变异,或是所有检测到的变异,还是应该包含全外显子组或全基因组序列?若是亲代基因组已经作为Trios家系策略的一部分进行了测序,那么家系数据应该如何以及在何处存储?数据访问是应该只限于参与直接服务于该家庭的临床医生,或也向工业界和/或学术界的研究人员开放?尽管具有基因组序列的个体在未来几年内可能在数据重复分析中获得临床益处,但是不断发展的测序技术可能意味着,如果临床指征明确,那么简单利用新的测序技术对其基因组进行重新测序,可能比起数据存储来说实际上更便宜、更简易且更具临床应用的信息价值。
数据的保密性与利用 NGS检测得到初步结果后,必须要在保护患者个体隐私和广泛数据分享以完善变异解读(为了患者个人或其他患者的利益)之间进行微妙地平衡。在儿科中,这种平衡更加复杂,父母经常被要求对孩子的基因数据做出决定,而这可能会造成不可逆转的影响。当孩子接近或超过法定年龄时,这些决定是否应被重新审视?关于谁能访问这些数据的问题,不仅涉及到诸如家庭成员、临床医生和研究人员等个人,还涉及到他们可获得数据的范围。问卷数据表明,父母对数据分享未来对他们的孩子带来风险的问题上,比成年患者更为关心。对数据的使用权限,可能限于特定诊断的基因变异,或是一系列临床相关的结果,也可能包括整个测序序列。对此,我们在之前提出过一种平衡数据深度和共享范围的比例方法。这种方法包括完全公开共享有限数量的潜在致病变异,从而最大限度地发挥诊断的潜在益处,同时最小化个体可能被重识别的风险。尽管共享个体全外显子组或全基因组数据也很重要,但个人被重识别的可能性要高得多,因此实际中经常使用具有严格管理访问权限的数据库来管理数据。共享匿名且有限的表型和基因型数据,不仅可以增加诊断和新发现的机会,而且还将增加极罕见病情的患者参与到新疗法的研究或临床试验的机会。
医疗服务的责任 对于临床医生来说,数据的访问问题与他们的医疗职责是否仅限于为患儿的直接问题进行诊断,或是否超出了初始检查的范围有关。这项医疗责任可以延伸到当新知识被揭示出来时,定期对数据进行再分析,有可能在最初测序的多年后才能对患者进行确诊,或者根据新证据来纠正之前的误诊。一些研究已经开创了一种定期更新基因panel进行迭代报告的方法,基于目前发现新基因的速度,这或许在儿科中产生实质性的诊断获益,尽管如此,但在临床中对基因组数据进行常规重新分析却受阻于用于更新和重新分析数据的资源不足以及需重新联系患者等情况。
医疗服务的职责还可以扩大到寻找倾向于成年发病的情况以及患儿及父母的不良药物反应的偶然发现。美国医学遗传学和基因组学协会推荐,应在检测数据中积极查询额外的“looked-for”的发现,尽管目前仅限于59个基因的致病变异。一般情况下,不建议对儿童进行无早期治疗的成人发病的疾病的额外查询,迄今为止,英国和欧洲的遗传学会一直在试图扩大基因组分析的范围,而非仅仅是寻求诊断。遗憾的是,解释无症状家族中可能的致病变异通常是困难的,因为大多数疾病基因已经在具有特定病症的个体或家系中被发现;因此,机会性基因组筛查(opportunistic genome screening)的益处和危害目前还不清楚。
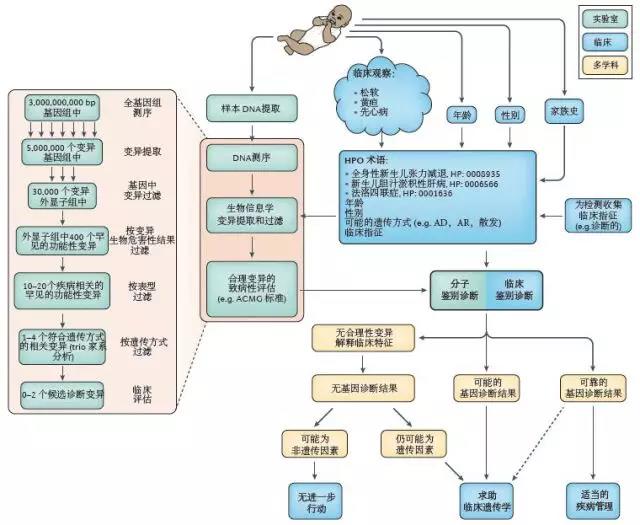
Figure 3 | 运用二代测序技术,整合临床和实验室的工作流程,用以优化罕见病的诊断
儿科基因组学服务的安全、有效的实施,需要精心的设计,以优化临床医师、生物信息学家以及科学家在诊断流程中各个部分的专业建议。变异筛选过程的每个步骤都必须谨慎考虑和充分评估,因为致病变异可能在任何步骤被疏忽筛除。尽管如此,仍需要严格的筛选使变异数量降到足够低的数量以供专家审核。通常次要等位基因频率MAF>1%或甚至>0.1%的变异会被筛除。需注意的是以下可能造成致病变异丢失的情况:选择了错误的基因转录本,在trios家系分析中误判了遗传模式(比如,表型轻微的父母被记录为表型正常),或者造成罕见病的变异类型在测序中或在检测流程中未能很好的捕获和检测(例如三碱基重复变异导致的疾病,如脆性X综合征)。
对患者的临床评估发生在诊断的开始和结束。在开始阶段,临床评估用于确定患者的临床表现存在可检测的遗传基础的先验可能性,以及用于确定临床表型和建立临床鉴别诊断。最后,临床医师应当评估实验室所提交的候选变异,能否构成明确的遗传病诊断(完整的或部分的诊断),以及诊断是否足够可信,得以指导患者未来的临床诊疗,并指导其亲属检测或胚胎植入前检测和产前诊断。在一些疑难病例中,罕见的候选变异常由临床专家和遗传专家组成的多学科小组会诊予以讨论(见FIG. 3)。ACMG,美国医学遗传学与基因组学学会;HPO,人类表型本体。(译者注:图示将流程划分为实验室和临床两部分,并且这二者是有机联系的,尤其是实验室需要基于临床表型信息进行分析,临床医生需要对基因数据结合临床进行评估。目前国内已有面向医生的诊断云平台系统,医生得以参与到检测报告数据的审核以及与实验室的互动沟通)
2.变异的过滤
关于将哪些变异纳入到诊断评估体系,哪些变异从后续分析中排除,在很大程度上取决于所使用的资源和访问日期。如果变异没有预测性的或已知的功能改变,或是被认为是太常见而不能成为特定疾病的原因,或者因为它们不符合所观察到的疾病的遗传模式,则这些变异通常会被排除(FIG. 3)。如果使用Trios模式, 则在对父母进行表型评估时需留意,以确保他们没有与患儿疾病相关的轻微的亚临床表型,否则会指向错误的遗传模式,从而混淆分析。过滤掉不具有已知功能的变异需要通过诸如Ensembl VEP 或 SNPeff 的软件准确预测基因变异的危害性影响。但是,这些预测可能并不能反映一个变异的真实的生物学危害性意义。此外,由于存在可变剪切体,相同的基因组变异可能具有不同的预测性结果,且缺乏一种简单的选择方法来确定大多数基因的临床相关转录本,这是不同诊断实验室之间差异的重要来源。识别常见变异需要访问具有大规模的人群资源数据信息的数据库,如ExAC数据库和gnomAD数据库。
在撰写该综述之际,这些数据库已包含了来自一系列成人研究队列的超过12 万个外显子组和15,000个基因组的变异数据,尽管许多种族人群数量仍然不足。导致严重儿童疾病的基因变异很少出现在成人疾病群体或其对照组的致病构型中,并且对早发型疾病来说,生活方式因素和长期的环境暴露不太可能导致其实质性的疾病多变性。因此,可以使用非常严格的等位基因频率阈值来筛选非常罕见的变异,用于患儿变异数据的进一步分析。然而,测序个体的种族以及其种族在参考数据库中的代表数据收录的情况,可能会影响变异过滤的有效性,且在基因组分析中仍然存在着欧洲偏倚(European bias)。
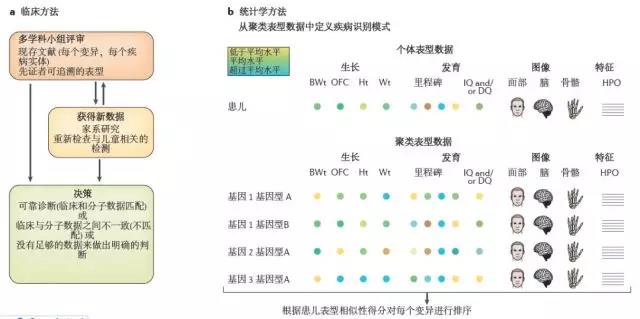
Figure 4 |基于表型的变异评级和解读的多学科小组评审
两种互补的方法(临床和统计学)可用于基于表型的变异评级。 a | 临床方法中,经过变异致病性、人群中出现的频率和遗传方式进行筛选后保留下来的变异,应参考患儿的临床表型来进行评估(译者注:即为生物、遗传、临床三要素分析)。这项工作最好在多学科专家小组的协助下进行,专家组对患者的临床表现、家族史、相关病史和其他检查结果(比如生化检查和影像结果)进行回顾,参考某个特定基因相关的临床特征的可用汇总数据(如 FIG. 4b) 和已报道的文献。分子诊断提示的候选结果可能需要新的数据(比如家系分析、进一步的临床检查或者对患儿信息的重新调研)来确定结论和进行可靠的基因诊断,或排除一个无法解释患者表型或家系遗传方式的基因变异结果。在部分患者中,变异是否致病并不能得到明确的结论。b | 表型匹配的统计学方法需要一定数量的在同一基因上存在致病变异的其他个体和目标患儿的分类的、定量的和/或基于影像的表型信息。这些数据可以用统计学方法对患儿表型与疾病模型匹配进行量化,并与其他不相关疾病的模型匹配进行比较分析。随着被基因诊断确诊的罕见和极罕见遗传病的患儿越来越多,这个方法将越来越可行。BWt,身体体重;DQ,发育指标;HPO,人类表型本体;Ht,身高;OFC,枕额周长/头围;Wt,体重。(译者注:BWt应为出生体重,Birthweight。应为原文错误)
3.致病性判断
临床检查是否全面详尽,对于解读基因组测序鉴定出的候选变异和保持良好的临床实践仍然是至关重要的(FIGS 3,4)。 对自动化流程筛选出的罕见变异进行致病性判断和确定基因诊断,是极具挑战性的。以往与所述疾病有关的基因变异通常通过设定基因panel进行优先考虑,并且鉴定出由非众所周知的致病病因导致的患者,有助于鉴定新的致病原因。
然而不幸的是,文献中常常出现假的基因-疾病关联关系,这导致了基因panel之间的巨大差异,而且关于依据何种证据水平将某个基因纳入一个基因panel中,尚缺乏明确的共识。此外,用于临床的致病性变异数据库,如Human Gene Mutation Database(HGMD),里面有大量错误,导致很多良性变异被错误的选中而形成貌似可信的诊断。随着基因组测序数据的增长,这种情形有可能会得到改善,但像gnomAD和ClinVar这类包含健康人群及罕见病人群的基因组大数据集,其重要性是不应被过度夸大的。儿科的大型队列研究,例如英国破译发育障碍性疾病的研究(Deciphering Developmental Disorders Study inthe UK),从统计学意义上建立了仅基于基因组数据判断致病性的可靠方法。该研究同时也参考了表型数据的分类和定量。分类方法使用结构化的分类法,例如人类表型本体(the Human Phenotype Ontology, HPO), 用以获得重要的临床特征,同时儿科遗传学的量化数据则包括生长发育关键节点的数据。
美国医学遗传学和基因组学学会-分子病理学协会(AmericanCollege of Medical Genetics and Genomics-Association for MolecularPathology, ACMG-AMP)指南 的出版是朝着建立变异分类的通用框架迈出的重要一步,且这一框架将持续被完善。DECIPHER之类的平台帮助临床医生进行标准化表型的捕获,并将其与基因型整合为单一资源内的患者级数据记录,从而能够对相关数据进行全面的探索分析。DECIPHER显示表型信息、成像数据、基因型信息(包括变异型,如SNV或CNV,危害性和遗传模式)以及2D/3D蛋白质结构(如果有的话),参考文献及种群信息等。它以ACMG变异致病性变异分类支持作为补充,并作为一种补充性的评估工具,促进基于分子和临床专业知识整合的多学科团队决策,从而实现可靠的基因诊断。系统性地捕获罕见和极罕见遗传病的患儿个体的分类和定量表型数据,也将有助于促进利用统计学方法来确定每种疾病的识别模式。这将使得在(历史数据中,译者注)已检测过的个体中所观察到的表型,与(当下案例中,译者注)由基因检测所产生的每种疑似致病基因型相关的所预期的表型之间,进行表型相似度的可靠的评估成为可能(FIG. 4b)。
4.数据共享
基因组数据的解读高度依赖于对对照群体和患者群体变异数据库的运用。然而,广泛的数据共享将面临后台维护的挑战——因其绝对庞大的数据量,和伦理方面的挑战——缘于不可能将基因组序列数据真正匿名化(BOX 1)。 通过DECIPHER 和 Phenome Central等资源共享组织化的或疑似表型变异的关联将促进这一进程,并最大限度地减少每个人共享的变异数量。诸如GA4GH Matchmaker Exchange 之类的举措,将这些数据与其他资源如(GeneMatcher) 联合起来,从而增加了搜索范围。ACMG最近发表了立场声明,即实验室和临床基因组的数据共享,对于改善遗传医疗保健至关重要。WES和WGS技术使我们能够在已知知识范围内进行罕见病的基因诊断,并可以通过仔细阅读当前的文献,掌握适当的注释平台和知识库的工作知识,以及与研究工作进行对接等方式加以不断巩固。理想状态下,这个框架应该由一个协作的基础设施来补充,以支持功能化和模式生物研究,从而带来更广泛的专业知识来实现可靠的分子和临床诊断。

关于儿科基因组学:在儿童罕见病诊断中的应用的后续内容更加精彩,请继续关注明日转化医学网……(转化医学网360zhyx.com)
 腾讯登录
腾讯登录

还没有人评论,赶快抢个沙发