【Nature子刊】窥探细胞内的秘密世界:浙江大学药学院程翼宇团队利用scCube,揭示转录组学中的多重变异性!
| 导读 | 本研究提出了scCube,这是一个Python包,用于对SRT数据进行独立、可重复和技术多样化的仿真。 |
近日, 浙江大学药学院程翼宇团队在期刊《Nature Communications》上发表了题为“Simulating multiple variability in spatially resolved transcriptomics with scCube”的研究论文。在本研究中,团队提出了一个空间分辨转录组学模拟器scCube,用于模拟空间分辨转录组学中的多个空间变异性,并生成无偏差模拟SRT数据。

https://www.nature.com/articles/s41467-024-49445-0
研究背景
01
空间分辨转录组学(SRT)技术的出现和快速发展,为在空间水平上了解组织的细胞组成、分子结构和功能细节,提供了前所未有的机会。各种新兴的尖端技术,包括基于成像的、基于空间条形码的下一代测序(NGS)和基于激光捕获显微切割 (LCM) 的方法,已成功应用于不同的生物学领域。例如,发育生物学、癌症和神经科学。
随着SRT数据的出现,用于下游分析的计算工具,也越来越先进。目前,科学界已经开发了300多个软件包,用于各种空间转录组的数据分析。例如,空间可变基因检测、细胞类型反卷积、未测量的基因插补票价、空间域识别和空间细胞的相互作用。虽然这些计算方法通常基于合理的假设,但如果没有金标准,就很难对其性能,进行基准测试和评估。
目前,一种广泛使用的评估计算方法,涉及从scRNA-seq数据构建的模拟SRT数据。然而,在scRNA-seq数据选择和用于在不同基准实验中,生成模拟SRT数据的方法方面,存在差异。在大多数情况下,模拟数据是专门基于所评估的计算方法的基础假设构建的,这可能会在评估过程中引入偏差。此外,大多数模拟步骤的描述,缺乏可重复性,可能会阻碍其他研究人员对这些方法的潜在重用。最近出现了几种生成合成SRT数据的方法。例如,SRTsim使用从参考数据推断的计数模型,模拟基因表达计数,然后将模拟计数,分配给合成数据中的空间位置。scDesign3应用了一个概率模型,该模型可以结合不同的细胞协变量,来模拟不同空间位置的基因表达变化。然而,SRTsim和scDesign3都基于SRT数据进行模拟,导致生成的数据,存在固有的局限性。例如,基因检测有限和细胞分辨率低。此外,scDesign3无法使用用户指定的新空间位置,模拟SRT数据,这极大地限制了其应用。然而,SRTsim可以根据参考数据中测量的\(k\)个最近相邻位置,为每个新位置分配模拟表达计数,但当参考数据和合成数据之间的组织形状或细胞类型空间分布,存在较大差异时,这种策略可能无法保留基因的空间表达模式。例如,Maynard等人的DLPFC数据,集中不同脑供体的切片。因此,仍然需要一种能够模拟独立、可重复、各种SRT数据的通用模拟框架,以更好地促进空间转录组数据分析方法的发展。
为此,我们引入了scCube,这是一种SRT模拟器,用于模拟空间分辨转录组学中的多个空间变异性,并生成无偏模拟SRT数据。基于变分自编码器(VAE)框架,scCube可以模拟scRNA-seq(或SRT)数据中不同细胞(或斑点)群体的基因表达谱。接下来,可以通过基于参考或无参考的策略,生成特定种群的空间分布模式。团队评估了scCube在各种真实SRT数据集上,使用现有单细胞或SRT模拟器的仿真性能,并展示了scCube在3个基准测试应用中的实用性。结果表明,scCube是一个用户友好的框架,可以模拟无偏差SRT数据,使研究人员能够更轻松、更准确地,对不同的计算方法进行基准测试和评估。
研究进展
02
使用scCube无参考策略模拟SRT数据中的多重变异性
团队将SRT数据的变异性,细分为两种类型:基因表达变异性和空间模式变异性。对于前者,scCube可以根据模拟的基因表达谱,选择用户指定的基因数量或类型,以模拟基于成像的SRT数据中靶基因的变异性。对于后者,scCube可以使用默认的空间自相关函数生成数量、类型和维度变化的细胞类型的模拟空间模式,并进一步模拟细胞类型(如细胞亚型)内空间模式的变异性,以及基于斑点的SRT数据中分辨率和斑点排列的变异性。团队通过以下3个示例,进行了详细的演示。
1.模拟细胞类型空间模式的变异性
scCube的一个基本功能,是它能够生成具有不同模式的细胞类型的空间分布。在scCube中,有两个重要参数\(\lambda\) 和 \(\delta\),它们分别控制生成的空间模式的模糊度和连续性。\(\lambda\) 的值,范围从0到1,较大的值,往往会形成更清晰的空间模式。因此,用户可以通过设置和组合不同的\(\lambda\)和 \(\delta\) 值,应用scCube,来生成特定的模式,以模拟不同组织中细胞的空间结构。
scCube的另一个功能,是它能够设置空间模式的数量,并仅模拟一组特定单元类型的空间模式。scCube的这一特性,使用户能够更好地模拟,真实组织中的不同情况。例如,特定细胞类型的空间域,或几种细胞类型的定位和分布。此外,用户可以应用此功能,来指定特定细胞类型,是否具有空间分布模式,从而实现手动指定该细胞类型,标记基因的空间表达模式类型。scCube生成的模拟三维SRT数据是无偏的,因此,足以作为基准数据集中的真实数据。scCube还提供了一个“拆分”功能,可以将三维SRT数据,拆分为用户定义的二维SRT数据,模拟数据的任意坐标轴。使用此功能,用户可以研究一系列相邻二维切片的空间变异性。例如,特定细胞类型的空间分布,及其标记基因的空间表达模式。通过联合利用模拟的三维SRT数据,和一系列推导的二维SRT数据,用户可以更准确地评估三维组织重建的计算方法。
2.模拟细胞类型内空间模式的变异性
scCube还提供了一个单独的功能,来考虑细胞类型内的异质性,并灵活地生成细胞亚型的空间模式。
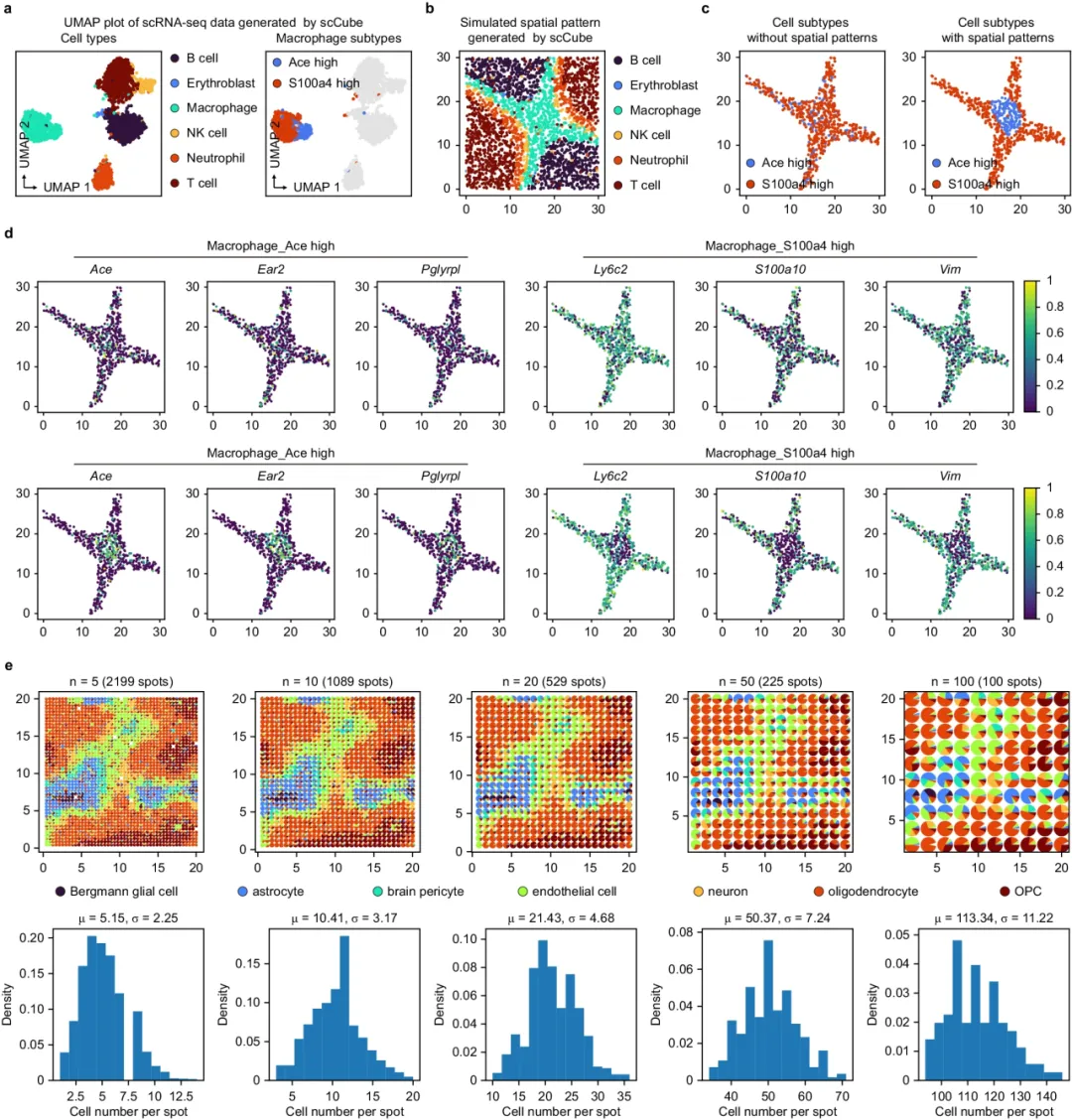
使用scCube模拟细胞类型内空间模式的变异性,以及基于斑点的SRT数据中,分辨率和斑点排列的变异性。
3.模拟分辨率和光斑排列的可变性
scCube还提供了,用于设置每个点的单元数(\(n\)) ,以及所有点的排列和邻域结构的可选参数。这使得在不同分辨率,甚至不同技术平台下,模拟基于点的SRT数据成为可能。
使用scCube模拟生物学上可解释的空间模式
在无参考空间模式模拟中,scCube进一步考虑,以自定义方式,生成更具可解释性的空间模式。用户可以首先模拟,一系列生物学上可解释的空间基础模式,包括非结构化的混合或成簇细胞群、围绕组织结构的细胞环,以及一些外部结构,如血管。然后,通过组合这些基本模式的不同类型和数量,灵活地生成高度定制的复杂空间模式。
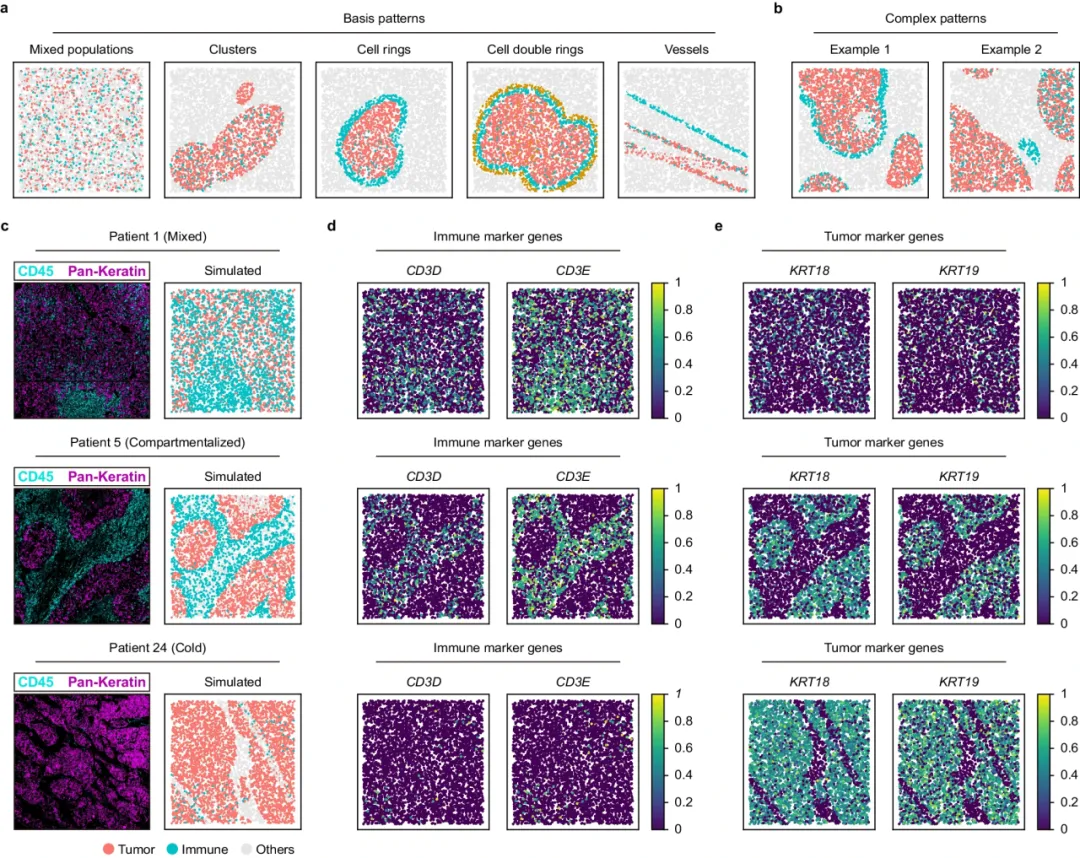
使用scCube灵活地模拟生物可解释的空间模式。
研究结论
03
在本研究中,团队提出了一个空间分辨转录组学模拟器scCube,用于模拟空间分辨转录组学中的多个空间变异性,并生成无偏模拟SRT数据。团队证明了scCube在真实SRT数据中,保留基因空间表达模式的能力;并说明了,scCube在对各种SRT分析计算方法,进行基准测试中的实用性,包括斑点反卷积、基因插补和分辨率增强。
scCube设计,包括SRT模拟的两个步骤:基因表达模拟和空间模式模拟。前一步主要基于VAE模型,该模型已成为单细胞组学数据建模的流行工具。团队还证明了VAE模型,可以准确地模拟原始数据的各种特征。例如,scRNA-seq或SRT数据的稀疏性。同时,团队提供了约300个经过训练的组织模型,这些模型与scCube Python包相结合,使用户能够更方便、更快速地,模拟真实组织中细胞的不同空间结构,而无需额外的训练步骤。scCube的一个潜在局限性,是其模拟的基于斑点的SRT数据提供的基本实况,仅限于细胞群水平。因为它在生成合成数据时,首先为每个斑点,分配一个细胞群。这可能不适合直接用于某些,以更精细分辨率运行的SRT分析方法的基准研究。然而,得益于scCube在细胞群注释选择中的稳健性,用户可以在模拟过程中,灵活选择适当粒度的细胞群注释。此外,由于scCube生成的基因表达谱,保持在斑点水平,用户还可以通过相关的下游分析(如斑点反卷积),细化每个斑点的注释粒度。
在空间模式模拟步骤中,scCube提供了两种策略:基于引用和无引用。基于参考的策略,使用SRT参考,旨在模拟基因跨位置的空间表达模式。来自不同测序平台生成的,不同组织的真实SRT数据的基准测试结果显示,scCube优于其他SRT或单细胞模拟器,具有广泛的可扩展性。此外,团队强调了scCube与SRTsim相比的独特优势,这是当前最先进的SRT模拟器。具体来说,scCube首先通过求解最优传输问题,在模拟数据中的单元(或点),与空间参考中的位置之间,构建映射。基于基因表达,然后将细胞(或斑点)映射到空间起源可能性最大的位置。该策略有效避免了组织切片形状、细胞类型比例和分布等外部因素的干扰,从而可以稳健地模拟基因的空间表达模式,无论是在相同切片,还是跨不同切片。相比之下,SRTsim在模拟中严重依赖空间参考的坐标系(即切片形状),当目标模拟数据的形状与空间参考的形状相同或足够相似时,SRTsim确实可以保留基因的空间表达模式。然而,当模拟与空间参考,存在较大差异的数据时,模拟的准确性会急剧下降。
对于无参考模拟,scCube旨在为细胞群,生成随机或定制的空间模式,并将其与模拟的基因表达谱相结合,从scRNA-seq数据重新构建完整的基准数据集。与SRTsim中实现的,从真实SRT数据开始的模拟相比,该策略可以生成模拟的SRT数据,该数据同时提供单细胞分辨率和整个转录组,这适用于评估一些整合方法(如斑点反卷积、基因插补和空间重建)的性能。在本研究中,团队通过设置指定的参数值,详细展示了scCube的无参考空间模式模拟的灵活性:包括基于随机方式的SRT数据中的多重变异性模拟,以及定制的更具生物学可解释性的空间模式的生成。
此外,scCube的一个理想特性,是它能够以用户指定的方式模拟SRT数据,以满足不同的基准测试目的;同时,提供真实数据中不存在的基本事实。具体来说,scCube允许在生成SRT数据时,改变一个特定变量。例如,基因的数量和类型,或空间模式的数量和面积,其允许用户灵活地研究,某些变量对不同计算方法性能的影响。团队还进一步讨论了,可能影响scCube生成的,模拟数据的方法性能的空间变异性。此外,对于3个应用中展示的SRT分析方法,scCube应该适用于其他计算工具的基准测试。例如,空间域识别和空间细胞-细胞相互作用推理方法。团队应用scCube对上述两种方法,进行了基准测试,并证明了空间信息有助于提高空间域识别,和空间近端LR对推理的准确性。综上所述,scCube能够提供可扩展、可重现、逼真的模拟,帮助用户更轻便、更准确地评估各种方法,更好地促进空间转录组数据分析方法的发展。
 腾讯登录
腾讯登录
还没有人评论,赶快抢个沙发