【Nature子刊】创新突破!上海交通大学俞章盛团队开发出快速、准确的空间可变基因鉴定方法
| 导读 | 本研究提出了HEARTSVG,这是一种无分布的、基于测试的方法,用于快速、准确地识别大规模空间转录组数据中的空间可变基因。 |
2024年7月7日, 上海交通大学俞章盛团队在期刊《Nature Communications》上发表了题为“HEARTSVG: a fast and accurate method for identifying spatially variable genes in large-scale spatial transcriptomics”的研究论文。通过对SVG进行聚类,团队揭示了人类结直肠癌数据中,两个不同的肿瘤空间域。其特征是,具有独特的空间表达模式、时空位置和生物学功能,从而揭示了肿瘤的复杂性。

https://www.nature.com/articles/s41467-024-49846-1
研究背景
01
空间转录组学,能够测量组织中的基因表达和位置信息。空间转录组学技术的发展,推动了组织结构的重建,并为发育生物学、生理学、癌症和其他领域,提供了深刻的见解。然而,空间转录组学(ST)数据的复杂性和高维性,对分析方法提出了新的挑战和要求。空间转录组学研究中的一个关键分析挑战,是鉴定空间可变基因(SVG)。其表达与空间位置相关,也称为SE基因(具有空间表达模式的基因)。识别SVG,有助于表征组织内的空间模式,并预测空间域。科学界已经开发了几种用于检测SVG的方法,其趋势是将数据建模为标记点过程,并测试成对点的空间分布和表达水平之间的显著依赖性。
Trendsceek、SpatialDE和SPARK,由于计算复杂度高,对大规模数据集的适用性有限。Trendsceek采用排列策略,来计算不同配对点的多个统计数据,这需要大量的计算工作,并且只能扩展到小规模数据集。在分析高维和稀疏ST数据时,高斯过程框架阻碍了SpatialDE和SPARK中SVG的检测和模型参数收敛。SPARK-X的计算速度,明显快于上述方法,但其有效性在很大程度上,取决于构建的空间协方差矩阵与真实底层空间模式的匹配程度。以上4种方法,通过搜索表达式和位置之间的预定义关系,来识别SVG。由于SVG真实空间模式的任意性,以及由此产生的表达式和坐标之间关系的不确定性,它们对各种空间模式的泛化性有限。scGCO能够识别具有未知确切位置和形状的SVG,但是,由于图切算法在识别SVG候选区域方面的准确性有限,尤其是在稀疏的ST数据集中,因此会出现假阴性。Squidpy的准确性,取决于随机排列的数量。增加随机排列的数量,可以提高结果的可靠性。然而,它的代价,是增加了时间消耗,使该过程更加耗时。
因此,团队建议使用HEARTSVG来克服这些限制,而无需提前了解有关SVG的信息或规范。团队采取相反的方法,识别非SVG,并使用此信息来推断SVG的存在。尽管基因表达与SVG的空间位置之间的关系尚不确定,但可以肯定的是,非SVG在基因表达与空间位置之间“没有关系”。HEARTSVG通过测试全局空间边缘表达式中的序列自相关,来识别非SVG。通过排除非SVG,其余基因被视为SVG。作为一种基于测试的方法,无需假设底层空间模式,HEARTSVG可以检测具有任意空间表达形状的SVG,适用于各种类型的大规模ST数据。团队进行了广泛的模拟,并将HEARTSVG方法,应用于由不同技术(包括10X Visium、Slide-seqV2、MERFISH和HDST)生成的12个真实ST数据集,以证明其准确性、有效性和计算效率。HEARTSVG在模拟中优于现有方法,具有更高的精度指标、计算效率和更低的误报(FP)。在分析真实的ST数据时,HEARTSVG在从不同空间转录组技术,获得的不同数据集中,识别具有不同空间表达模式的具有生物学意义的SVG。HEARTSVG具有扩展到包含数百万个数据点的数据集的潜力,并为研究SVG,提供了一系列精心设计的分析工具,从而能够解开复杂的生物现象。
研究进展
02
HEARTSVG识别SVG并预测空间域
10X Visium是癌症研究中,应用最广泛的商业空间转录组学技术。团队将HEARTSVG应用于人类结直肠癌(CRC)数据集,其使用10X Visium技术生成,涉及4,174个斑点和15,427个基因。团队对这个数据集,进行了无监督聚类和细胞类型注释,并结合了之前的研究信息染色(H&E)组织图像。这种组织,包含5种主要的细胞类型:肿瘤细胞、平滑肌细胞、正常上皮细胞、固有层和成纤维细胞。肿瘤细胞,位于两个不同的区域。HEARTSVG鉴定的SVGs,表现出显著的生物学相关性。与其他5种方法相比,HEARTSVG在19个肿瘤相关的KEGG通路(包括癌症:概述和信号转导)中,显示出更小的P值和更大的基因交叉大小。HEARTSVG表现出最高的AUC(AUC = 0.843,分别为0.727),强调了其生物学可解释性。
对于已识别的SVG,团队利用自动聚类模块,预测了6个主要空间域,并对每个空间域中的SVG,进行了富集分。一些空间域,与特定的细胞类型相关,与无监督空间聚类结果一致。
团队将HEARTSVG,应用于来自同一队列的,另外两个结直肠癌ST数据集和相应的肝转移ST数据集。HEARTSVG 的 AUC (平均 AUC = 0.792) ,高于其他方法。在6个结直肠癌和肝转移空间转录组学(ST)数据集中,与结直肠肿瘤样本中的非肿瘤区域相比,团队检测到肿瘤细胞中,许多线粒体编码基因的表达更高。然而,在肝转移样本中,没有观察到这种现象。团队认为,结直肠癌原发部位的肿瘤细胞,比转移性肝癌部位具有更高的氧化磷酸化(OXPHOS)活性。总体而言,HEARTSVG成功检测到,具有视觉上不同模式的SVG。自动聚类模块,基于位于细胞类型内外的可区分的SVG模式,有效地预测了空间功能域。

a 原始苏木精和伊红染色(H&E)组织图像(左)和无监督空间聚类结果(右)。HE图像中的红色圆圈区域,代表肿瘤区域。b 气泡图显示了不同方法中,19条肿瘤相关通路(x 轴)的KEGG通路富集分析结果。每个气泡代表一个通路,其大小对应于每种方法检测到的通路和SVG的重叠基因大小。图的x轴和y轴,表示不同的方法及其重要性(\({-\log }_{10}({{{\rm{p}}}}-{{{\rm{value}}}})\))。P值由 gProfiler计算。c ROC曲线,说明了使用肿瘤微环境的共同基因模块(左)和结直肠癌亚型的共识分子标记物(右),作为真实SVG的金标准时,6种不同方法的TPR和FPR。d HEARTSVG基于SVG预测4个空间域,并绘制每个空间域中SVG的平均表达。e 具有代表性的SVG,对应于Fig. 3d中的4个预测空间域。f 富集分析,对应,4个预测的空间域。柱线的长度,表示使用 \({-\log }_{10}({{{\rm{p}}}}-{{{\rm{value}}}})\)的丰富。P值由 g:Profiler 计算。
HEARTSVG识别具有空间模式的标记基因
团队分析了由多路复用错误鲁棒荧光原位,杂交产生的小鼠视前下丘脑的2个数据集。MERFISH能够对单个细胞进行空间分辨的RNA分析,具有高精度和高效性。通过MERFISH生成的数据适度稀疏,在超过一半的细胞中,检测到超过40%的基因。第1个数据集涉及6,112个细胞和155个基因,由8种细胞类型组成。第2个数据集,由10种细胞类型组成,涉及5,665个细胞和161个特征(156个基因和5 个空白对照)。HEARTSVG、SpatialDE、SPARK、SPARK-X、scGCO和Squidpy,在第1个MERFISH数据集中,鉴定了133、154、149、141、65和145个基因。在第2个MERFISH数据集中,鉴定了128、161、145、132、46和144 个基因。所有方法的结果,高度一致。然而,SpatialDE错误地将5个空白对照,归类为具有顶级基因等级的SVG。HEARTSVG、SPARK、SPARK-X和Squidpy,报告了1个空白对照组的误报,等级较低;scGCO没有报告假阳性。scGCO遗漏了一些具有清晰空间表达模式的SVG。
在这2个数据集中,HEARTSVG有效地识别了,与空间上位于特定区域的细胞类型相关的SVG。团队利用自动聚类模块,来获取多个空间域。由此产生的空间域,始终与其相应的细胞类型匹配。例如,在第1个数据中,团队分别预测了,对应于少突胶质细胞和兴奋性神经元的2个空间域。总体而言,自动聚类模块,突出了 HEARTSVG软件的实用性。
HEARTSVG在各种数据集中具有普遍适用性
为了评估HEARTSVG的通用性,团队将其应用于更全面的数据集,包括由高清空间转录组学(HDST)生成的小鼠嗅球数据和使用10X Visium的2种不同癌症的ST数据集。HDST数据集巨大而稀疏,由181,367个斑点和19,950个基因组成,超过98%的斑点检测到的基因,少于50个。只有HEARTSVG、SPARK-X、scGCO和Squidpy,可以完美地对HDST数据进行操作,并分别检测到447、89、0和248个SVG。scGCO无法识别此稀疏HDST数据集中的任何SVG。HEARTSVG鉴定了排名靠前的SVG(Gm42418、mt-Rnr1、mt-Rnr2、Cmss1、Gphn)。这些SVGs,显示出明显的空间表达模式,尽管在如此稀疏的数据中,观察基因的视觉空间表达模式,具有挑战性。
10X Visium是癌症研究中最流行的ST技术。因此,团队分析了10X Visium生成的其他ST数据,包括原发性肝癌 (PLC)ST数据集,以及伴脑转移肾透明细胞癌(RCC-BM)ST数据集,旨在展示HEARTSVG的卓越性能。与以前的应用一致,这些数据集中的肿瘤细胞,表现出复杂性和高度异质性,包括同一组织内,具有不同功能的多种肿瘤细胞类型。HEARTSVG有效地识别了癌症ST数据中与肿瘤相关的SVG,并预测了几个具有不同功能的空间域。例如,PLC ST数据,包含3种不同的肿瘤细胞类型。通过HEARTSVG鉴定肿瘤相关SVG,并预测其相应的空间结构域,揭示了这些细胞类型之间的潜在协同功能。此外,团队发现,许多肿瘤相关基因,在肿瘤的“小巢”中比在“大巢”中,表现出更高的表达。SVG检测,有助于进一步了解肿瘤间和瘤内遗传异质性,以及复杂的肿瘤微环境(TME)和癌症机制,这对于了解肿瘤进展和对治疗的反应,至关重要。
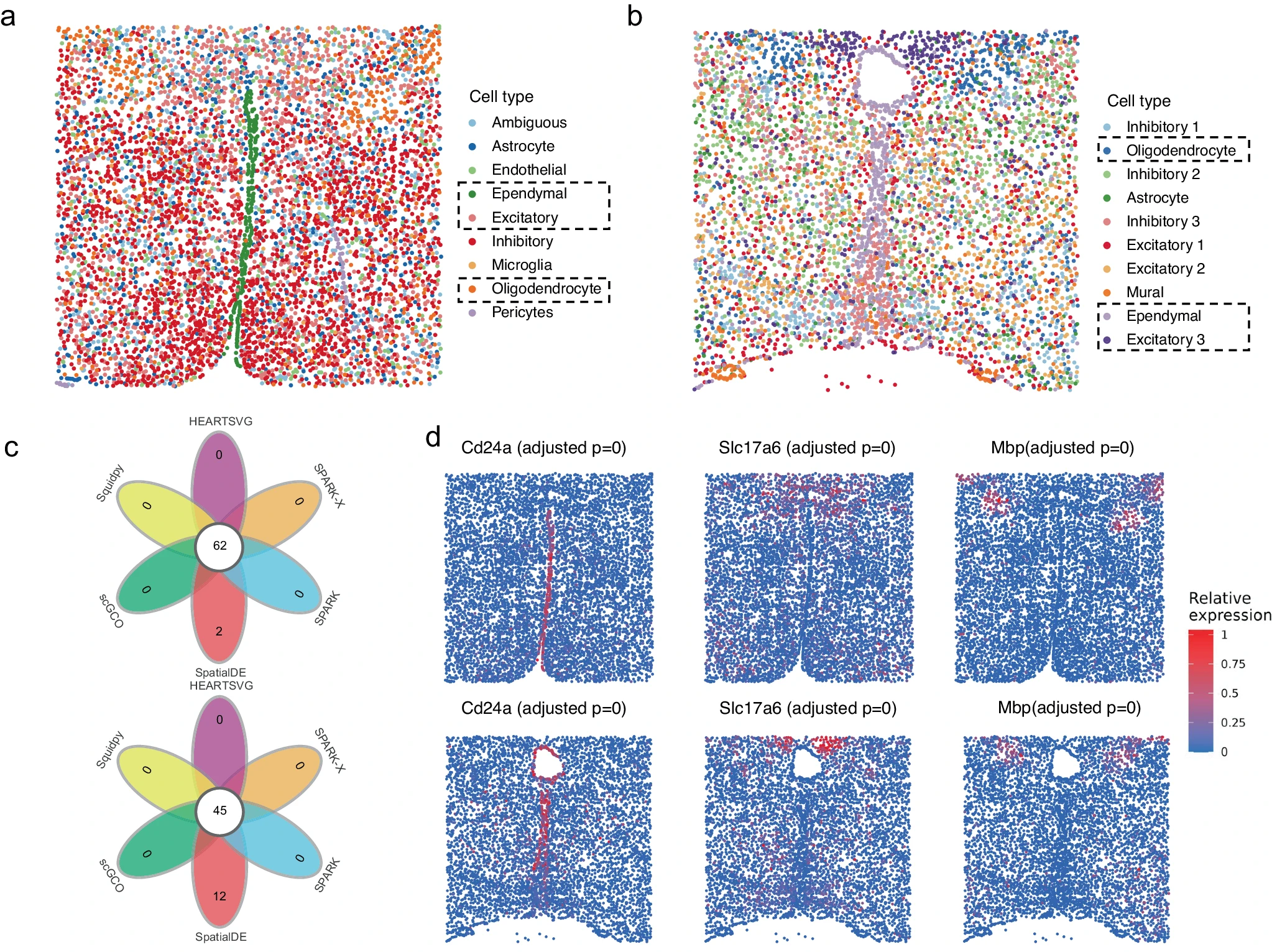
a MERFISH数据1(6,112个细胞,155个基因)的已知细胞注释的可视化。b MERFISH数据2(5665个细胞,156个基因和5个空白对照)的无监督空间聚类结果的可视化。c 由HEARTSVG、SpatialDE、SPARK、SPARK-X、scGCO和Squidpy识别的SVG维恩图。d 室管膜、兴奋性和少突胶质细胞类型的代表性标记基因的可视化,以及HEARTSVG在2个 MERFISH数据集中,获得的相应调整后的P值。P值由HEARTSVG计算,并使用Holm方法进行调整。图5c和图5d的上面板,对应于MERFISH数据1。下面板,对应于MERFISH数据 2。
研究结论
03
团队提出了HEARTSVG,一种基于测试的无分布方法,用于快速、精确地,检测大规模空间转录组数据中的 SVG。与现有的SVG检测方法不同,HEARTSVG使用另一种策略,通过排除非SVG基因,来推断SVG的存在,使其能够在来自不同空间技术的各种ST数据集中,以高精度、有效性和泛化性,识别任何空间表达模式的SVG。得益于测试框架和缺乏底层数据生成模型,HEARTSVG具有卓越的计算效率和可扩展性,非常适合大规模空间转录组学数据。此外,HEARTSVG 软件还提供了用于SVG高级分析的各种功能,包括自动聚类、富集分析和可视化工具。
团队的研究评估了HEARTSVG在模拟和真实ST数据上的性能,证明了其在各种情况下的准确性、稳健性和通用性,包括不同数量的细胞、SVG标记面积的百分比、空间模式和空间转录组测序技术。HEATSVG在大多数仿真场景中,具有最高的\({F}_{1}\)分数,并且具有良好的可扩展性和计算效率。HEARTSVG、SPARK-X、scGCO和Squidpy,能够在100万个细胞的数据集上,成功运行。然而,HEARTSVG和SPARK-X,表现出比 scGCO和Squidpy更低的时间消耗。scGCO实现了出色的误报率控制,但由于候选区域识别的不准确性,其性能会因在稀疏模拟数据集中,忽略大量SV而受到阻碍。其他研究也揭示了scGCO在SVG鉴定方面的局限性。在SVG百分比增加的模拟数据集中,SPARK-X的误报率增加,而HEARTSVG的误报率,保持在较低水平。此外,HEARTSVG可以检测出具有不同空间格局的SVG,而SPARK-X在识别SVG时,具有模式偏好,难以检测出一些非条纹图案和小比例的SVGs标记区域。
团队在3种不同组织(结直肠、肝脏和大脑)的4种不同空间转录组测序技术(10X Visium、Slide-seqV2、HDST 和 MERFISH)的12个数据集上,实施了 HEARTSVG。HEARTSVG 表现出最高的 AUC(平均 AUC = 0.792),证明了其在具有不同数据特征的数据集中的准确性和有效性。在本研究中,团队对3种不同类型癌症(结直肠癌、原发性肝癌和肾细胞癌脑转移)的ST 数据集进行了分析,这些数据集是使用10X Visium生成的,这是一种广泛用于癌症研究的商业ST技术。肿瘤的ST数据,包含很少的细胞类型,其SVG主要与肿瘤细胞相关。HEARTSVG(8个重要通路)、SPARK-X(8个重要通路)、scGCO(7个重要通路)和Squidpy(7个重要通路)在10X Visium结直肠癌数据中,鉴定出比SpatialDE(2个重要通路)和SPARK(2个重要通路)更多的癌症相关KEGG通路。此外,HEARTSVG软件的SVG自动聚类模块,有助于预测具有不同空间表达模式的不同肿瘤相关空间域。在结直肠癌ST数据中,肿瘤细胞位于样本的两个非相邻区域。团队发现,两个肿瘤相关空间结构域,仅在一个肿瘤细胞区域,具有高表达模式,而不是两个区域。基于SVG的空间域预测,揭示了肿瘤错综复杂的功能多样性和超越细胞分类的协同相互作用,为肿瘤组织的生物学复杂性,提供了新的视角。
总体而言,HEARTSVG是一种检测空间可变基因的强大方法,能够识别任意形状的空间表达模式。此外,在HEARTSVG软件中加入自动聚类模块,增强了对生物过程的理解,展示了HEARTSVG在空间转录组学数据分析中的多功能性和潜力。但是,HEARTSVG存在仅依赖空间坐标的局限性。在未来的研究中,将基因表达与相应的H&E组织图像相结合,结合H&E组织图像的信息,将更全面地了解疾病进展中的细胞机制。
 腾讯登录
腾讯登录

还没有人评论,赶快抢个沙发