【Nature子刊】北京医院张瑞团队:首次揭示真实世界多中心转录组测序的研究结果!
| 导读 | 作为Quartet项目的一部分,团队在45个实验室中进行了一项RNA-seq基准研究,系统地评估了真实世界的RNA-seq性能,并研究了26个实验过程和140个生物信息学管道中涉及的影响因素。 |
2024年7月22日,北京医院张瑞团队在期刊《Nature Communications》上发表了题为“A real-world multi-center RNA-seq benchmarking study using the Quartet and MAQC reference materials”的研究论文。通过同时基于Quartet和MAQC样本的质量评估,本研究彻底阐明了真实世界RNA-seq的性能,特别是在检测细微差异表达时。

https://www.nature.com/articles/s41467-024-50420-y
研究背景
01
转录组测序(RNA-seq)为探索全球表达模式,以及识别可变剪接事件,拓宽了新途径。转录组学数据的差异表达分析,能够全基因组鉴定与感兴趣的生物学条件相关的基因或亚型表达变化。这极大地促进了疾病诊断生物标志物的发现、预后和治疗选择。这些证据有助于RNA-seq在临床常规中的应用。
过去10年中,社区中RNA-seq的质量评估,主要依赖于具有里程碑意义的MAQC参考样本。其特点是样本之间的生物学差异显著,这些样本是由微阵列/测序质量控制(SEQC/MAQC)联盟,从10个癌细胞系(MAQC A)和23个供体的脑组织(MAQC B)中开发的。MAQC联盟利用这些样本和来自外部RNA对照联盟(ERCC) 的92个合成RNA的加标,来评估RNA-seq性能,并展示了在适当的数据处理和分析条件下,跨不同位点和平台的相对表达测量的高精度和可重复性。
值得注意的是,基于MAQC参考物质的质量控制,可能无法完全确保准确识别临床相关的细微差异表达。此外,与之前研究设计中严格控制的RNA-seq工作流程相比,真实世界场景在样本处理、实验方案、测序平台和实验室分析管道方面存在显著差异,其中混杂因素可能会影响RNA-seq的准确性和可重复性。因此,有必要对真实世界RNA-seq在检测临床诊断目的的细微差异表达方面的整体性能,以及影响诊断性能的技术因素,进行详细的质量评估。
在Quartet项目的范围内,本研究利用带有ERCC对照加标的Quartet RNA和MAQC RNA样本,在45个独立实验室中生成RNA-seq数据,且每个实验室都使用自己的内部实验协议和分析管道。通过同时基于Quartet和MAQC样本的质量评估,本研究彻底阐明了真实世界RNA-seq的性能,特别是在检测细微差异表达时。本研究为RNA-seq的实验和生物信息学设计,提供了最佳实践建议,并通过比较Quartet和MAQC参考样本,强调了在细微差异表达水平上,进行质量控制的必要性。
研究进展
02
实验设计的最佳实践
实验执行质量低下,会显著影响RNA-seq的性能。来自16个实验室的RNA-seq数据,未能通过多项质量指标,被认为在样品库制备或测序过程中,实验质量低下 。值得注意的是,这些低质量的实例与实验方法的选择无关,因为它们分布在各种实验工作流程中。因此,团队的研究结果,强调了多维质量控制在实验设计中的重要性。
不同的实验方案往往会影响RNA-seq的性能,在实验设计时应予以考虑。(i)在表达数据质量方面,Poly(A) 选择方法的SNR值,高于rRNA 去除方法。(ii)对于绝对表达水平,团队观察了mRNA富集方法和链性的影响,其中,rRNA耗竭法和链特异性文库与参考数据集的相关系数更高。(iii) 对于相对基因表达和DEG水平,映射到外显子区域的更多reads数,显示出强或中等相关性。
团队还观察到,基于单一类型的基本事实,使用不同RNA输入、样品库试剂盒和测序平台的实验室,在准确性方面,存在统计学上的显著差异。此外,对于插入片段大小(被认为是影响基因或亚型鉴定和定量的因素),团队的研究结果表明,在其他因素的参与下,它与基因定量准确性的相关性较差。
除了基因定量方面外,某些实验因素(如测序深度和读取长度),也被认为会影响外显子连接检测,这意味着对亚型和可变剪接鉴定有影响。即使较低的测序深度,也足以检测已知的连接;并且,增加测序深度,进一步促进了新连接的鉴定。
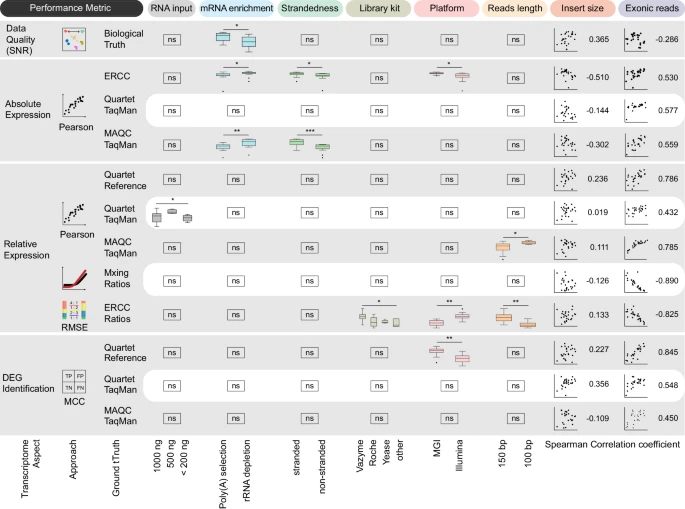
基于不同性能指标的实验因素的影响。
生物信息学设计的最佳实践
与RefSeq注释相比,Ensembl始终导致更高的唯一映射率和更低的多重映射率。STAR总体映射率最高,且具有独特映射率。STAR要么映射,要么丢弃配对读段,避免了未配对的单端读段的对齐。HISAT2和Subread具有相当的独特映射率,但HISAT2在大多数样本中,往往具有略高的多映射率,导致总体映射率更高。Subread对接受错配的容忍度更高,主要集中在较少的错配碱基上。Ensembl注释更为复杂,导致验证了更多数量的交汇点。对于这些已知的交界点,两种对齐工具没有表现出显著差异,而HISAT2确定了更全新的交界点。大多数新的结点都不可靠,在应用基于计数的阈值后,数量显著减少。
在选择RefSeq基因注释时,无比对定量工具表现出更高的准确度,尤其是Salmon,其性能与基因组比对定量流程相似。RSEM表现出中等准确度,优于结合Ensembl注释的免比对量化工具。总体而言,团队基于4种基本事实,对相对基因表达测量的所有定量管道的性能排名,支持选择Ensembl基因注释(或使用StringTie时的RefSeq注释)和基因组比对定量策略,进行基因定量目的。
选择平衡TPR和精度的阈值,似乎是一种有效的方法,但缺乏用于评估灵敏度或精度的基准数据集,在实践中是一个挑战。相比之下,计算最大DEG数是实用的。尽管基于最大DEG数和最高TPR的阈值之间,存在细微差异,特别是在Quartet样本中。
基因组比对定量工具,始终产生更高的DEG鉴定准确度。Salmon在无比对定量工具中是一个例外,其显示出与外显子水平定量工具相似的更高准确度。RSEM对Quartet样本的准确度较低,但对MAQC样本的准确度较高。
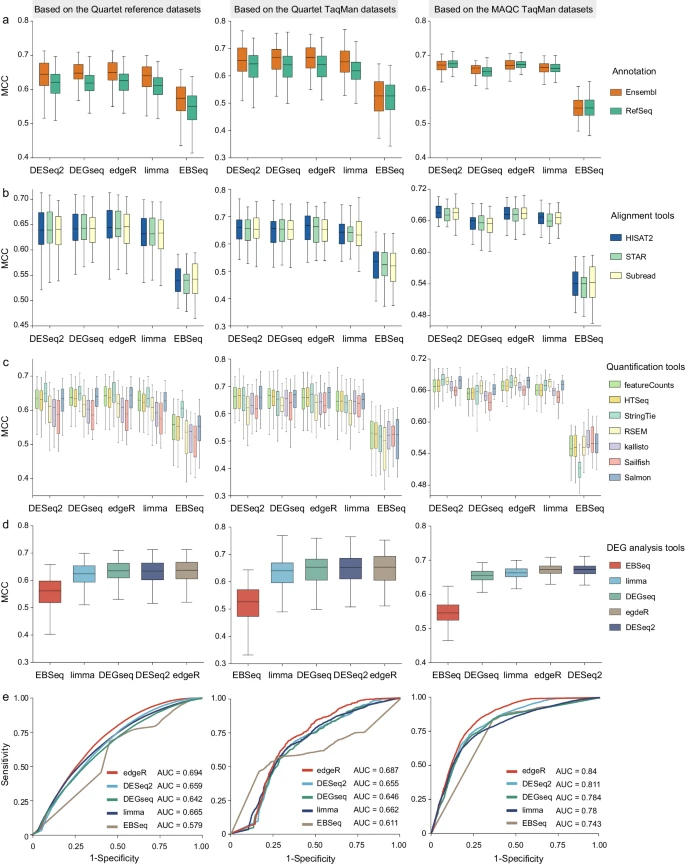
每个生物信息学步骤,对DEG鉴定准确性的影响。
研究结论
03
本研究首次揭示了转录组分析性能在现实世界中的显著差异,特别是在检测Quartet样本时,细微的差异表达。首先,团队从多个角度检查了数据质量,共识别出16批低质量数据,这似乎比之前报道的更常见。其次,绝对表达水平在实验室间表现出显著的差异,而相对表达检测更可靠,这已得到广泛认可。第三,DEG的数量差异很大,DEG调用的准确性指标表明,即使专注于蛋白质编码基因,实验室的准确度指标也存在广泛的差异。因此,团队的研究结果强调了这样一个事实,即真实世界的RNA-seq性能,可能无法完全满足临床诊断需求,需要持续改进质量,特别是针对细微的差异表达。
与MAQC样品(38.2%和34%)相比,实验和生物信息学过程中的技术因素,导致Quartet样本(85.1%和75.1%)的绝对基因表达水平,变异比例更高。对于Quartet和MAQC样本,相对表达将技术因素引起的变异,减少到20%以下,表明其消除批量效应的重要性。
总之,这项研究揭示了真实世界转录组谱中的显著实验室间差异,特别是在检测细微的差异表达、在数据质量、绝对表达和差异基因表达方面。在实验和生物信息学方面,对实验室间变异来源的研究,突出了RNA-seq方法开发和优化的关键点。本研究为RNA-seq的实验和生物信息学设计,以及质量控制,提供了最佳实践建议。这些将帮助研究人员准确识别疾病状况的细微变化,加速RNA-seq向诊断工具的转变。此外,这些数据还可用于解决转录组谱分析的其他方面,包括选择性剪接、基因融合、RNA编辑和RNA变异。
参考资料:
1.Stark, R., Grzelak, M. & Hadfield, J. RNA sequencing: the teenage years. Nat. Rev. Genet 20, 631–656 (2019).
2.Habgood-Coote, D. et al. Diagnosis of childhood febrile illness using a multi-class blood RNA molecular signature. Med 4, 635–654.e5 (2023).
 腾讯登录
腾讯登录

还没有人评论,赶快抢个沙发