【Nature子刊】中山大学李伟忠团队运用深度学习方法,探秘癌症特异性代谢
| 导读 | 在本研究中,团队介绍了DeepMSProfiler,这是一种可解释的基于深度学习的方法,能够对原始代谢信号进行端到端的分析,并具有高精度和可靠性的输出。 |
2024年8月20日, 中山大学医学院李伟忠团队在期刊《Nature Communications》上发表了题为“An end-to-end deep learning method for mass spectrometry data analysis to reveal disease-specific metabolic profiles”的研究论文。团队开发的端到端深度学习方法DeepMSProfiler,为疾病诊断和机制发现,提供了一种简单可靠的方法,增强了其广泛的适用性。

https://www.nature.com/articles/s41467-024-51433-3
研究介绍
01
代谢组学提供了生物系统中小分子浓度的全面视图,在发现用于诊断目的的疾病生物标志物方面,发挥着关键作用。然而,代谢组学在精准医疗中的广泛应用,可能会受到诸如复杂数据处理、高批次间变异性和繁重的代谢物鉴定等障碍。
深度学习已在各种组学数据分析中得到广泛应用,有望解决代谢组学数据的复杂性。然而,深度学习需要高质量的数据和足够数量的样本,否则,会导致诸如预测模型过度拟合等问题。由于深度学习方法通常被认为是“黑匣子”过程,因此,在生物医学研究的背景下,模型可解释性对于预测的重要性,越来越得到认可。
在本研究中,团队开发了一种集成端到端深度学习方法,称为基于深度学习的质谱分析器(DeepMSProfiler),用于非靶向代谢组学数据分析。DeepMSProfiler有效地消除了不同医院之间不良的批次效应和变化,并推断出与特定分类相关的未注释代谢物。它还利用集成模型策略,优化来自多个单个模型的特征归因。在独立测试数据集中,DeepMSProfiler的受试者工作特征曲线下面积(AUC)得分为0.99,在检测早期肺腺癌方面的准确率为96.1%。
研究进展
02
肺腺癌、良性结节和健康个体的代谢组学特征
为了分析肺腺癌、良性结节和健康个体之间的整体代谢差异,团队从DeepMSProfiler中提取了RISE统计的特征贡献热图。通过将标签信息映射到热图上,团队能够定位对应于不同m/z和保留时间的代谢物,从而获得其特征贡献分数。在真阳性健康和良性结节样本中,贡献最大的代谢信号均匀地位于200至400m/z之间,并在1-3分钟内。相比之下,位于200至600m/z之间,以及1-4分钟内的代谢信号在肺腺癌样本中贡献最大,但其他地区的信号也具有相对较高的分数。
团队将这些m/z信号与通过串联质谱(MS2)鉴定的代谢物,一起输入到基于蛋白质-蛋白质和蛋白质-代谢物相互作用的分析工具PIUMet中,以构建与疾病相关的特征网络。肺腺癌样本中成功匹配了82种蛋白和121种代谢物。因此,该分析允许发现与疾病状态相关的未知代谢信号,尽管当前模型的分辨率在区分有助于疾病分类的所有单个峰方面,可能相对较低。热图和通路分析一致表明,健康个体和良性结节,具有相似的代谢特征。相比之下,癌症组呈现出独特的特征,具有特定的通路,并且与健康个体或良性结节共享通路中的代谢物或蛋白质数量增加。综上所述,网络和路径分析,证明了基于深度学习的DeepMSProfiler的可解释性。

使用基于LC-MS的非靶向血清代谢组的DeepMSProfiler方法。
模型在结肠癌中的应用
在结肠癌数据集的独立测试数据集中,模型实现了97.9%(95%CI,97.7%–98.1%)的准确度, 98.7%(95%CI,98.6%–98.8%)的精确度, 召回率为93.4%(95%CI,92.9%–94.1%), F1为95.8%(95%CI,95.4%–96.2%)。这些结果表明,DeepMSProfiler具有出色的可转移性。
泛癌中代谢蛋白网络的发现
DeepMSProfiler构建了一个包含23个不同类别的模型,然后从23个类别的模型中提取特征,以识别每个类别各自的关键代谢信号。用于可解释分析的最终集成模型,达到了99.3%的准确度、97.2%的灵敏度和100%的特异性。
团队成功地生成了疾病特异性代谢物-蛋白质网络,以及贡献分数热图。其中,贡献分数超过0.70,被认为是疾病特异性代谢物的指示。团队确定了14种代谢物和3种蛋白质,它们在23种癌症相关代谢物-蛋白质网络中,表现出共现。团队将代谢数据与甲基化信息进行关联,验证了PLA和UGT基因家族与高贡献疾病特异性代谢物之间的关联。以前的研究已经报道了PLA和UGT基因家族在多种疾病中的重要作用,如PLA2G7和PLA2G6在乳腺癌、前列腺癌和神经退行性疾病,以及UGT3A2在头颈癌中的重要作用。这些证据支持了DeepMSProfiler的发现。总之,跨越泛癌情景的扩展分析,突出了DeepMSProfiler在发现潜在疾病相关代谢物和蛋白质方面的能力。
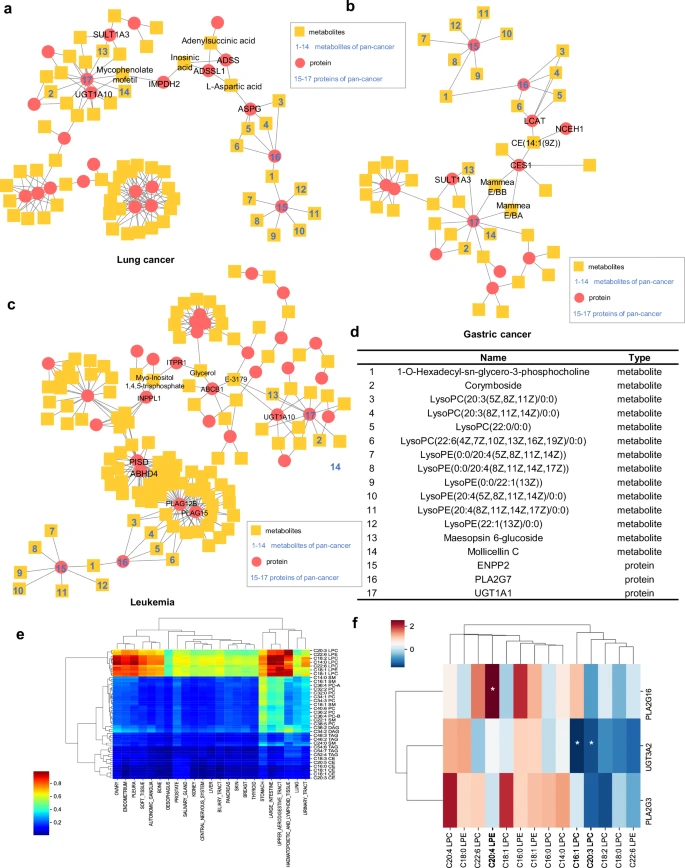
23种癌症类型的代谢物和蛋白质关联。
研究结论
03
在本研究中,团队采用深度学习方法在LC-MS代谢组学分析中捕获原始信号,而不影响数据完整性。团队利用基于网络的分析工具PIUMet,进一步实现了从m/z到通路注释的直接过渡,有效鉴定了癌症组的82种蛋白质和121种代谢物,而MS2注释的代谢物为9种。
此外,该方法能够覆盖通过常规注释识别的代谢物,同时揭示未检测到的疾病特异性特征。通过采用端到端策略,团队揭示了有助于每组不同代谢组学特征的完整生物学输出。例如,在肺腺癌的特征中,确定了色氨酸的代谢特征。其结果与之前通过常规注释方法的发现一致,即与良性结节和健康对照组相比,早期肺腺癌中色氨酸通路中的代谢物减少。丝氨酸和甘氨酸通过介导一碳代谢对核苷酸合成也很重要,这与靶向非小细胞肺癌的治疗策略有关。团队还观察到胆汁分泌在肺腺癌概况中的贡献,这与浸润性肺腺癌中胆汁酸代谢异常的另一份报告一致。需要注意的是,模型的分辨率可能受到限制,无法区分所有有助于疾病分类的单个峰。
DeepMSProfiler生成的高分辨率热图,显示了对预测类别的特征贡献,以及特定代谢组学信号的精确位置。它能够提供可解释的分析,以确保研究人员预测的生物学合理性。凭借批量效应去除、全面的代谢组学分析和集成策略的能力,DeepMSProfiler在不同类别中表现出一致且稳健的性能。它对肺腺癌、良性结节和健康样本的预测AUCs超过0.99,对早期(I 期)肺腺癌的准确率为96.1%。此外,它能够揭示肺癌之外的潜在疾病相关代谢物和蛋白质。
总之,DeepMSProfiler提供了一种简单可靠的方法,适用于疾病诊断和机制发现,有可能推动代谢组学在精准医疗中的应用。其有效的端到端策略,应用于原始代谢组学数据,可以在疾病筛查和诊断的非侵入性临床实践中,使更广泛的人群受益。
参考资料:
1.Schmidt, D. R. et al. Metabolomics in cancer research and emerging applications in clinical oncology. CA Cancer J. Clin. 71, 333–358 (2021).
2.Li, H. et al. The landscape of cancer cell line metabolism. Nat. Med. 25, 850–860 (2019).
 腾讯登录
腾讯登录

还没有人评论,赶快抢个沙发