【Nature子刊】强强联合!西湖大学合作南方医科大学共同发现细胞命运的调控新机制
| 导读 | 导读:细胞命运决定涉及基因组的重新连接,但在染色质水平上仍然知之甚少。 |
近日,西湖大学裴端卿和南方医科大学朱平共同在国际知名期刊《Nature Communications 》发表题为“The NuRD complex cooperates with SALL4 to orchestrate reprogramming”的研究论文,该研究报道了染色质重塑复合物NuRD在体细胞重编程的早期阶段参与关闭开放染色质。Sall4,Jdp2,Glis1和Esrrb可以有效地将MEF重新编程为iPSCs,但只有Sall4是必不可少的,能够招募NuRD的内源性成分。然而,与已知的Sall4-NuRD相互作用相比,敲低NuRD组件只能适度地减少重编程。
值得注意的是,这些缺陷可以通过将NuRD相互作用的基序嫁接到Jdp2上来部分挽救。对染色质可及性动力学的进一步分析表明,Sall4-NuRD轴在重编程的早期阶段在关闭开放染色质中起着关键作用。在由Sall4-NuRD闭合的染色质位点中,编码抗重编程的基因。总之,该研究表明NuRD在重编程中以前未被认识到的作用,并可能进一步阐明染色质闭合作为细胞命运控制的关键步骤。

https://www.nature.com/articles/s41467-023-38543-0
研究背景
01
细胞命运决定涉及基因组的重新连接,但在染色质水平上仍然知之甚少。在这里,研究人员报道了染色质重塑复合体NuRD在体细胞重编程的早期阶段参与关闭开放染色质。Sall4、Jdp2、Glis1和Esrrb可以有效地将MEF重编程为iPSCs,但只有Sall4能够招募NuRD的内源性成分。然而,与通过突变或删除NuRD的n端相互作用基序来破坏已知的Sall4-NuRD相互作用(使Sall4无法重编程)相比,敲除NuRD组件只能适度地减少重编程。值得注意的是,通过将NuRD相互作用基序嫁接到Jdp2上可以部分修复这些缺陷。染色质可及性动力学的进一步分析表明,在重编程的早期阶段,Sall4-NuRD轴在关闭开放染色质方面起着关键作用。在被Sall4-NuRD关闭的染色质位点中,编码了抗重编程的基因。这些结果确定了NuRD在重编程中以前未被认识到的作用,并可能进一步阐明染色质关闭是细胞命运控制的关键步骤。
研究发现
02
研究表明,JGES重编程与经典的山中因子OSKM明显不同,因为JGES依赖于Sall4参与内源性NuRD关闭mef中的开放染色质,而OSKM依赖于mefs10,60中的开拓因子打开染色质。Sall4-NuRD轴可能在正常发育和疾病过程中发挥类似的作用。
虽然具有相同的起始细胞,即mef和最终结果,即能够嵌合体形成和种系传播的iPSCs,但JGES可能与OSKM协调重编程完全不同,因此,提供了一个难得的机会来比较和对比它们在介导细胞命运决定方面的策略。基于OSKM结合转录因子通常无法接近的染色质的能力,已经提出了一种开创性的重编程模型。人们一直认为这种开创性功能是OSKM重编程的一个关键特征,这些因子通常不在mef中表达,因此它们的结合基序大多以封闭的形式隐藏在染色质中。早期的研究表明,在重编程的早期阶段,OSK激活内源性因子,如Sap30,这些因子将参与Sin3A关闭mef中打开的染色质。通过确定Sall4-NuRD轴,研究人员提出类似的染色质关闭事件应该是细胞命运转变的起始事件。
本发现似乎与先前的多项研究相矛盾,这些研究认为NuRD亚基在重编程中是一个负调节器,其中一项研究发现Gatad2a和Chd4缺失导致iPSC衍生效率高达100%。本研究在许多方面与其他研究不同。在本重编程系统中,MEF被JGES逆转录病毒感染进行重编程,他们选择了转基因的“二次重编程”胚胎成纤维细胞(mef),携带teto诱导的OKSM诱导iPS。本研究报道了OKSM和7f诱导的mef (Jdp2, Esrrb, Sall4, Nanog, Kdm2b, Mkk6, Gkis1)在7天的过程中遵循不同的分子轨迹到达最终naïve状态。
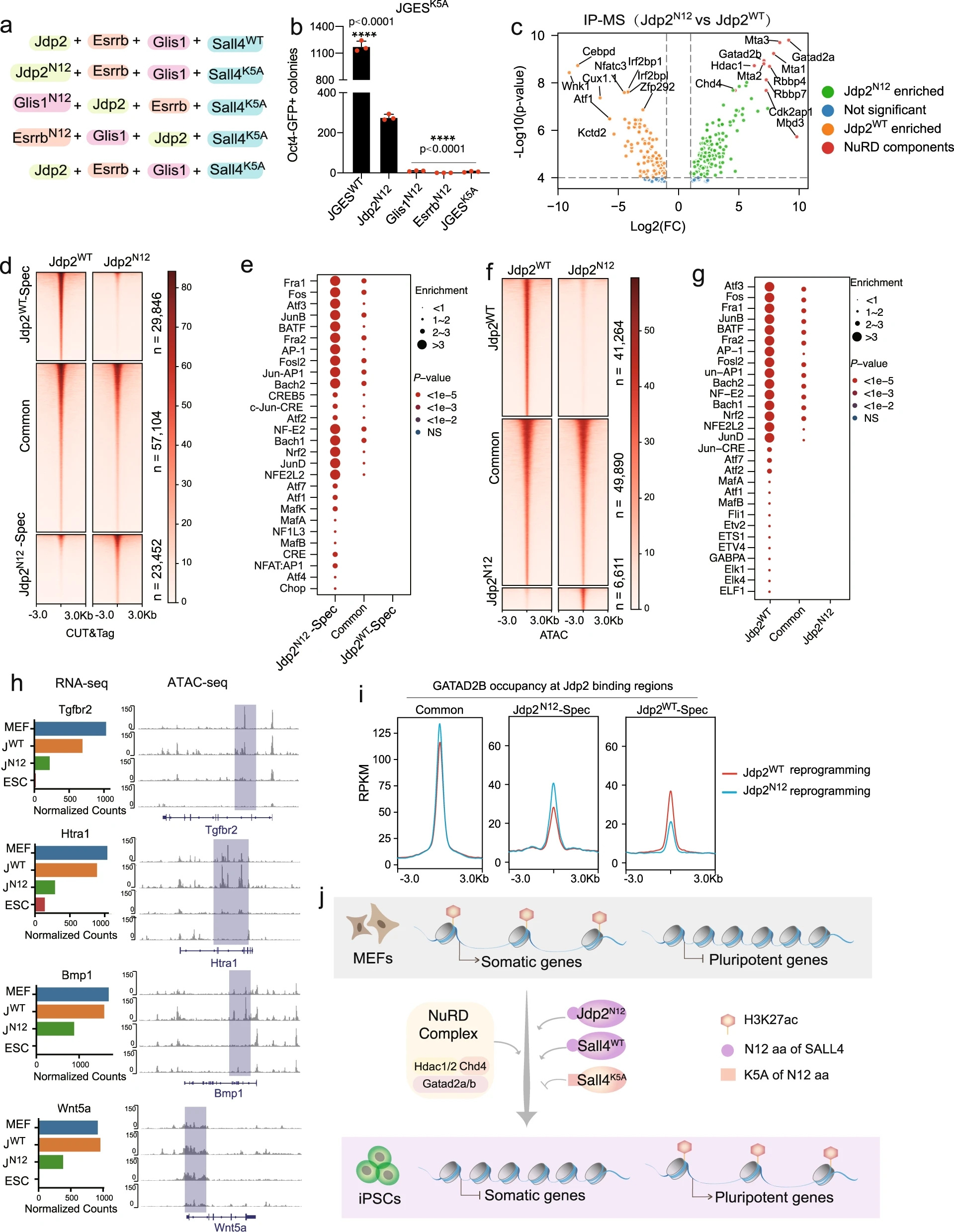
NuRD 复合体与 SALL4 合作编排重编程
研究意义
03
总之,研究结果表明,NuRD复合物通过关闭MEFs中的染色质在重编程的早期阶段发挥积极作用。(转化医学网360zhyx.com)
参考资料:
https://www.nature.com/articles/s41467-023-38543-0
注:本文旨在介绍医学研究进展,不能作为治疗方案参考。如需获得健康指导,请至正规医院就诊。
 腾讯登录
腾讯登录


还没有人评论,赶快抢个沙发